7 Padrões Pontuais I
7.1 O que são Padrões Pontuais?
A análise de padrão de pontos é o tipo mais simples de análise de dados espaciais. Baseia-se na localização dos eventos em um determinada área de estudo a partir das coordenadas. O objetivo é estudar a disposição espacial dos pontos a partir de suas coordenadas.
Os processos pontuais são definidos como um conjunto de pontos cuja localização está em \(R^2\), sendo esse processo gerado por um mecanismo estocástico.
Os pontos são os pares de coordenadas (x, y), que representam os eventos (observações, indivíduos, lugares ou qualquer outro objeto discreto definido no espaço).
| Evento | Coord X | Coord Y |
|---|---|---|
| 1 | 4,30 | 2,45 |
| 2 | 5,39 | 3,35 |
| 3 | 4,10 | 3,50 |
- Em geral o que se deseja é verificar a ocorrência de padrões espaciais nos pontos e se estão aglomerados espacialmente (clusters) ou se estão aleatoriamente distribuídos .
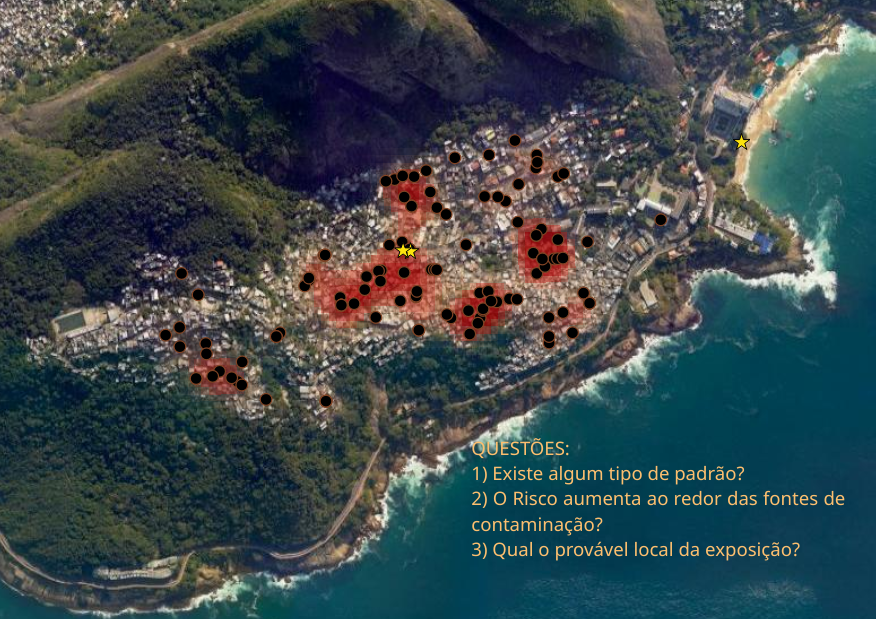
No exemplo abaixo temos os pontos dos casos suspeitos em uma investigação de surto de hepatite A no Rio de Janeiro no verão de 2017/2018. As estrelas amarelas mostram locais onde foi encontrado o vírus da hepatite A (HAV) em amostras de água.
7.2 Tipos de Distribuições
Aleatória: qualquer ponto tem a mesma probabilidade de ocorrer em qualquer local e a posição de qualquer ponto não é afetada pela posição de qualquer outro ponto;
Uniforme: cada ponto é tão longe de todos os seus vizinhos quanto possível;
Cluster: Muitos pontos estão concentrados juntos, e grandes áreas podem conter pouquíssimos pontos, se houver algum.
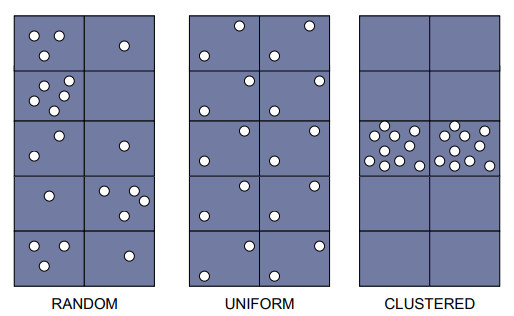
Fonte: Geospatial Training Workshop
Lendo os bancos com as localizações dos casos de homicídios, suicídios e acidentes de carro em Porto Alegre/RS.
As principais bibliotecas do R que estaremos usando nos exemplos abaixo são:
sfspatstatsplancs
library(tidyverse)
library(spatstat)
library(sf)
library(splancs)
# lendo os bancos
local <- "https://raw.githubusercontent.com/ogcruz/dados_eco_2023/main/dados/"
homic <- read.table(paste0(local, "homic.dat"), col.names = c("x",
"y"))
suic <- read.table(paste0(local, "suic.dat"), col.names = c("x",
"y"))
acid <- read.table(paste0(local, "acid.dat"), col.names = c("x",
"y"))
# Plotando os casos de homicídios em um plano
# cartesiano
g <- ggplot(homic) + geom_point(aes(x, y, color = "Homicídios"),
shape = 1) + labs(color = "")
g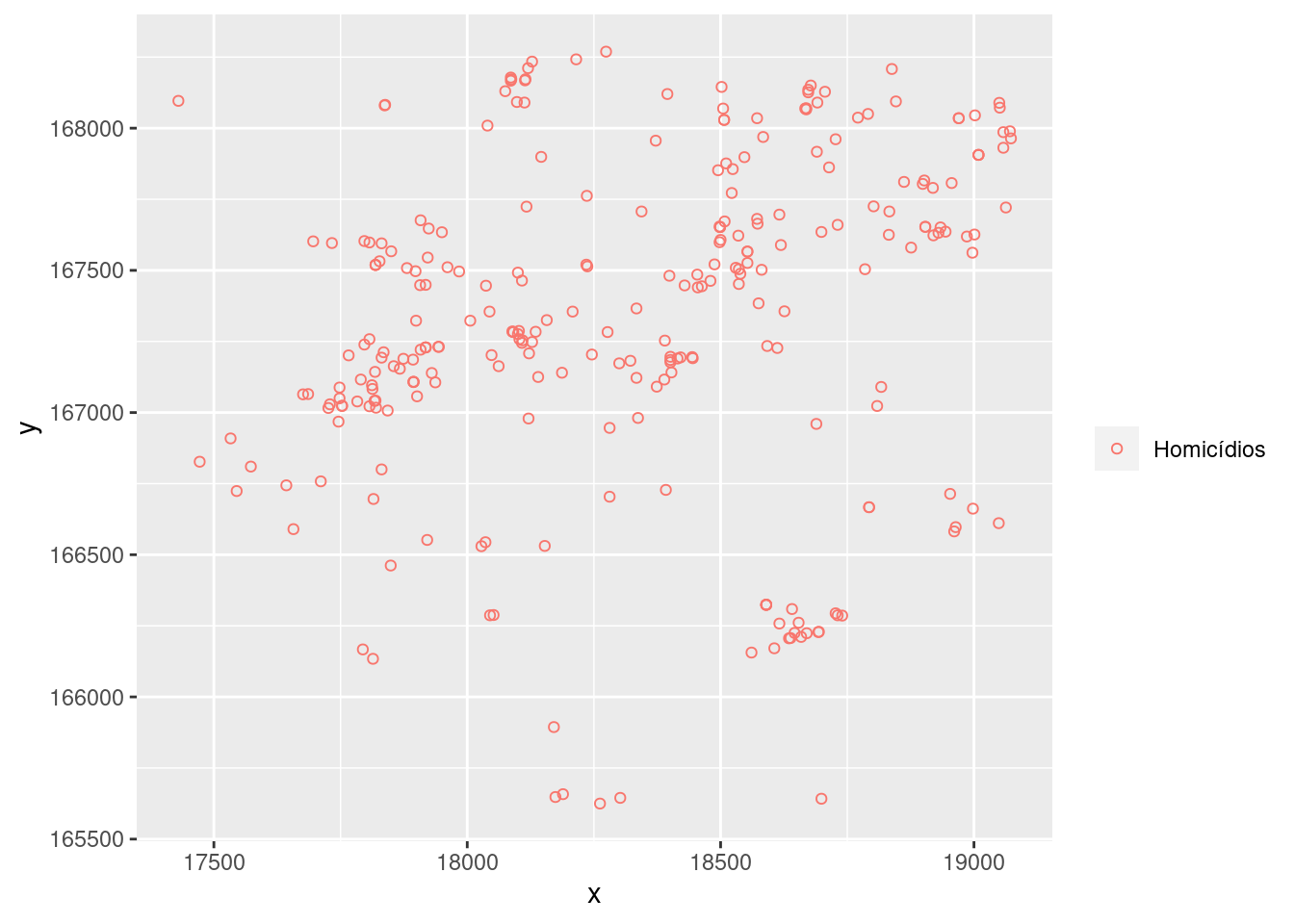
Porto Alegre é uma cidade disposta ao longo do eixo norte/sul. O grafico perdeu a estrutura espacial, ajustando para o tamanho e forma da janela. Por isso é necessario informar ao programa que este tipo de objeto é um objeto espacial e tem uma escala, que deve ser preservada. Vamos fazer isso com transformando-o em um objeto espacial do pacote sf.
# Transformando os pontos em geometria espacial
# de pontos
homic_sf <- homic %>%
st_as_sf(coords = c("x", "y"))
suic_sf <- suic %>%
st_as_sf(coords = c("x", "y"))
acid_sf <- acid %>%
st_as_sf(coords = c("x", "y"))
# Repetindo o mesmo gráfico. Repare que agora
# usamos a função `geom_sf()`:
g <- ggplot() + geom_sf(data = homic_sf, aes(geometry = geometry,
color = "Homicídios"), shape = 1) + labs(color = "")
g
Agora vamos adicionar os pontos referentes a suicídios e acidentes:
g <- g + geom_sf(data = suic_sf, aes(geometry = geometry,
color = "Suícidios"), shape = 1) + geom_sf(data = acid_sf,
aes(geometry = geometry, color = "Acidentes"),
shape = 1)
g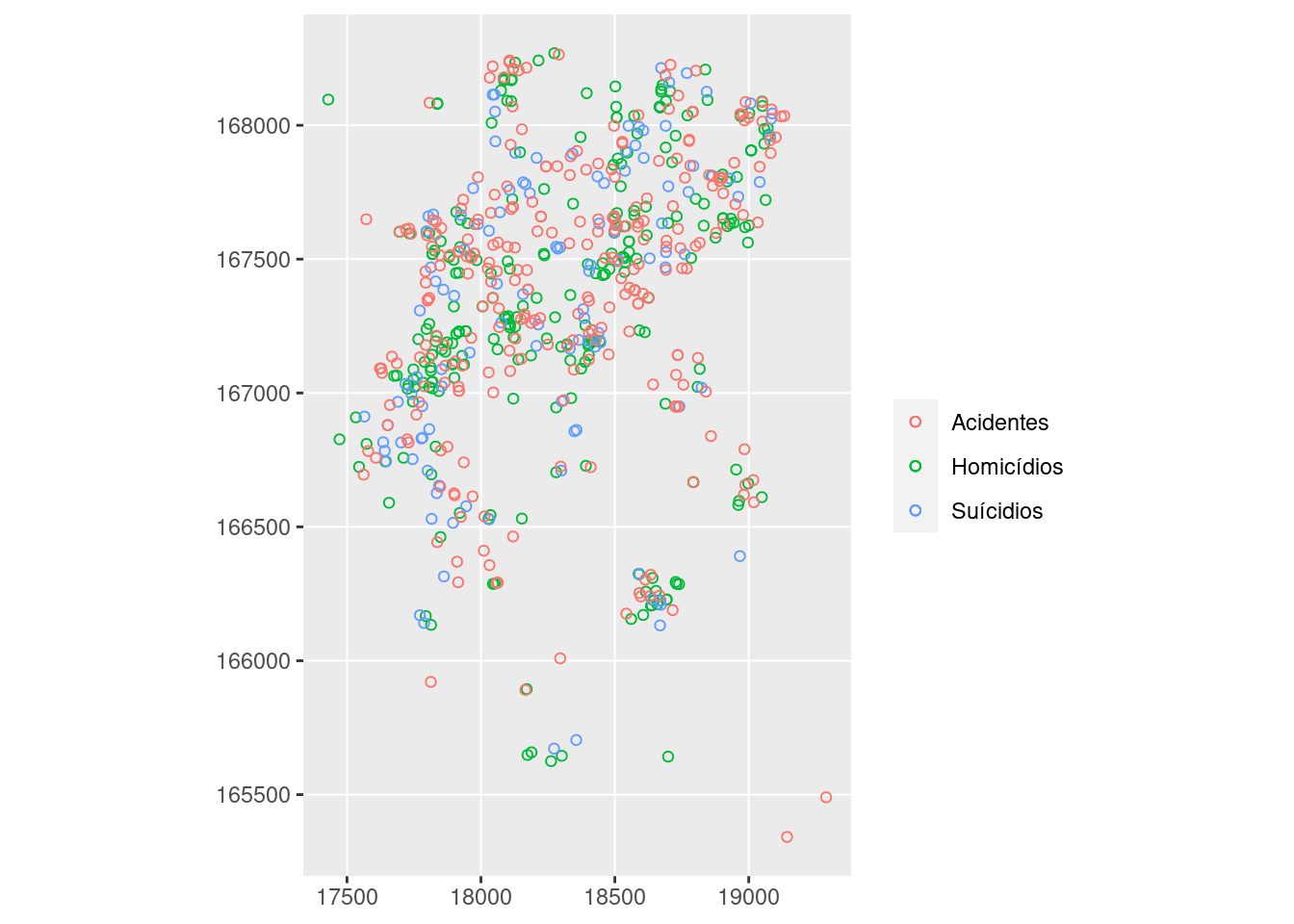
Importando o polígono do contorno de Porto Alegre:
# contorno de porto alegre
contorno.poa <- read.table(paste0(local, "/contpoa.dat"),
col.names = c("x", "y"))
# plotando com a função `geom_polygon`:
g + geom_polygon(data = contorno.poa, aes(x, y), fill = "transparent",
color = "black")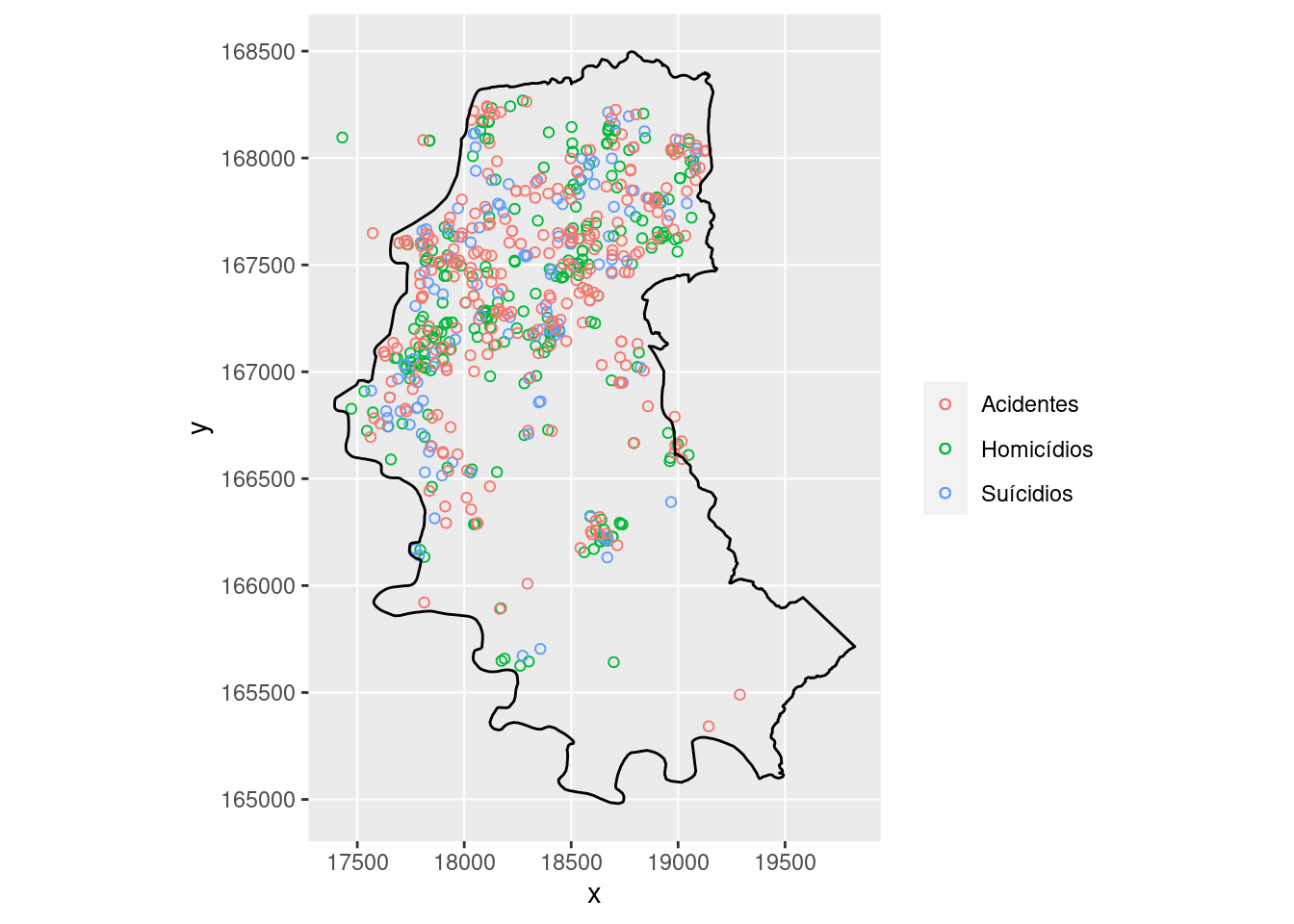
Os pontos fora do contorno sao das ilhas, não devem ser incorporados a análise. Vamos transformar o contorno também em um objeto espacial (st_polygon) para identificar os pontos fora do contorno:
library(sf)
# Para transformar em polígono espacial,
# transformamos primeiro em matriz e em seguida
# em uma lista:
poa_sf <- contorno.poa %>%
as.matrix() %>%
list() %>%
st_polygon()Agora sim, podemos utilizar a função st_within() para identificar os pontos fora do contorno:
# A função st_within() tem essa funcionalidade
homic_sf <- homic_sf %>%
mutate(dentro = lengths(st_within(homic_sf, poa_sf)))
ggplot(homic_sf, aes(geometry = geometry)) + geom_sf(aes(color = as.factor(dentro))) +
geom_sf(data = poa_sf, fill = "transparent") +
labs(color = "Dentro do polĩgono")
7.3 Processos pontuais
Os processos pontuais podem ser descritos como:
- Processo de primeira ordem, considerados globais ou de larga escala, que correspondem a variações no valor médio do processo no espaço.
- Tal processo pode ser representado por mensurações da intensidade baseado na densidade dos pontos (média dos eventos) na área de estudo (ex: Estimativa de Kernel).
- Processo de segunda ordem, denominados locais ou de pequena escala, é o processo representado pela interação entre dois pontos arbitrários.
- O objetivo desse processo é a mensuração da dependência espacial baseado na distância entre os pontos (ex: Vizinhos mais próximos, Função K).
7.4 Completa Aletoriedade Espacial (CSR - complete spatial randomness)
\(H_0\): Os pontos estão distribuidos aleatoriamente no espaço
\(H_1\): Os pontos podem formar clusters ou estão dispersos no espaço
CSR assume que os pontos seguem um processo homogêneo de Poisson na área de estudo.
Simulando alguns padrões dos dados de ponto, temos o seguinte:
- Simulando os processo espaciais:
O pacote sf possui a função st_sample() que permite a geração de \(n\) pontos dentro de uma janela:
set.seed(9999)
# padrão aleatório
alea_sf <- st_sample(poa_sf, size = 100, type = "random")
g_alea <- ggplot(alea_sf, aes(geometry = geometry)) +
geom_sf() + geom_sf(data = poa_sf, fill = "transparent") +
ggtitle("Distribuição aleatória")
g_alea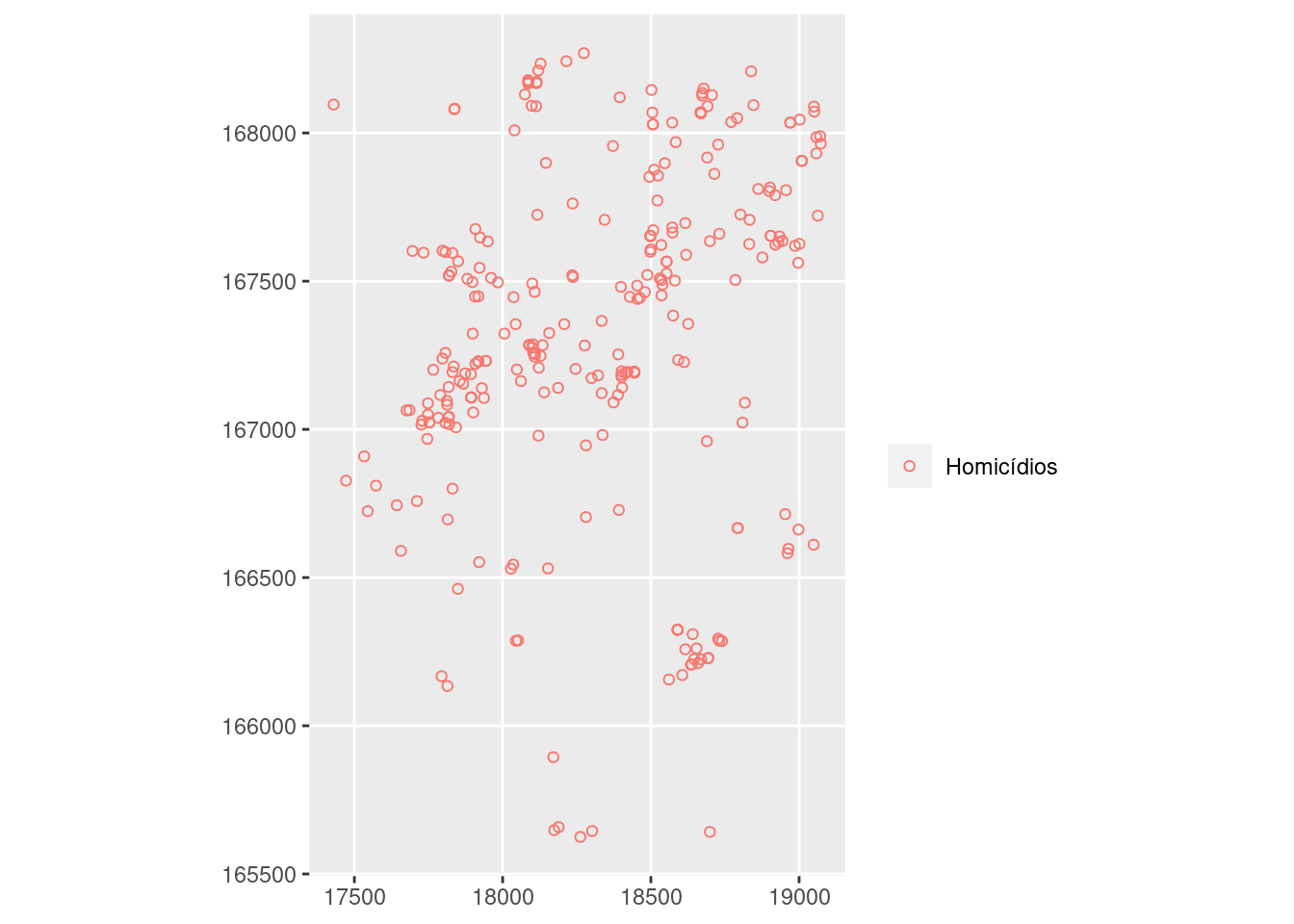
# padrão regular
uni_sf <- st_sample(poa_sf, size = 100, type = "regular") %>%
st_as_sf() %>%
# a função jitter é bastante utilizada e
# serve para adicionar uma flutuação (desvio)
# aleatória aos pontos
st_jitter(amount = 100) %>%
filter(lengths(st_within(., poa_sf)) == 1)
g_uni <- ggplot(uni_sf, aes(geometry = x)) + geom_sf() +
geom_sf(data = poa_sf, aes(geometry = geometry),
fill = "transparent") + ggtitle("Distribuição regular")
g_uni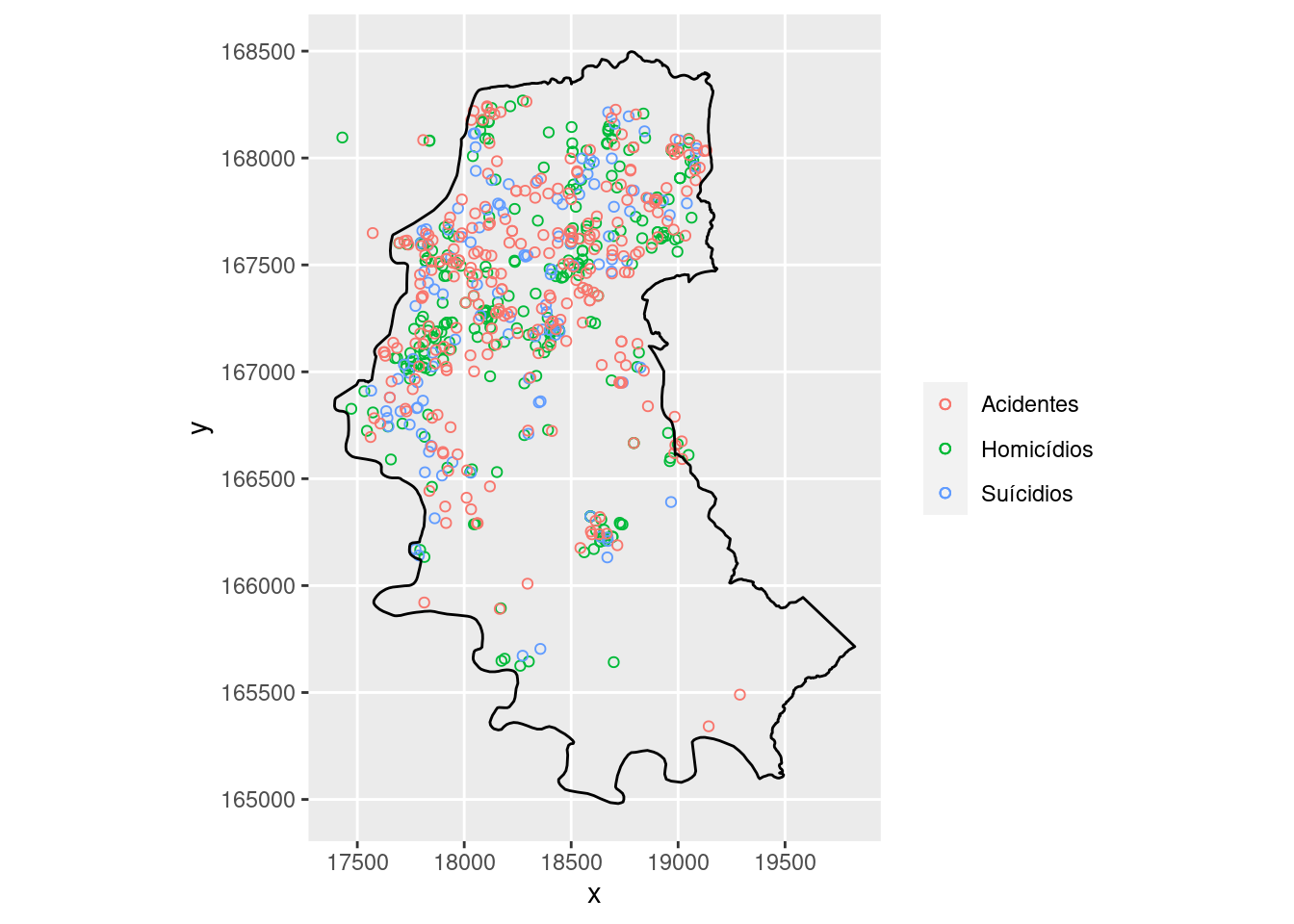
A função st_sample() permite a geração de pontos clusterizados a partir de um processo determinado, como um processo de cluster de Poisson, ou processo Thomas (para mais informações, ver ??spatsat.random). Essas funções levam um tempo para gerar as amostras, por isso iremos implementar uma função mais simples:
# Função para gerar uma distribuição
# clusterizada: Recebe um conjunto de centroides
# dos clusters e gera pontos ao redor deles
st_make_clusters <- function(centroids, n, amount,
factor = 0.002) {
clustered <- centroids # começa com centroides dos clusters e adiciona pontos em volta
for (i in seq_len(n)) {
clustered <- clustered %>%
rbind(centroids %>%
st_jitter(amount = amount, factor = factor))
}
clustered
}
# agora sim, gerando um padrão clusterizado
clu_sf <- st_sample(poa_sf, size = 10, type = "random") %>%
st_as_sf() %>%
st_make_clusters(10, amount = 200, factor = 0.5) %>%
filter(lengths(st_within(., poa_sf)) == 1)
g_clu <- ggplot(clu_sf, aes(geometry = x)) + geom_sf() +
geom_sf(data = poa_sf, aes(geometry = geometry),
fill = "transparent") + ggtitle("Distribuição aglomerada")
gridExtra::grid.arrange(g_alea, g_clu, g_uni, ncol = 3)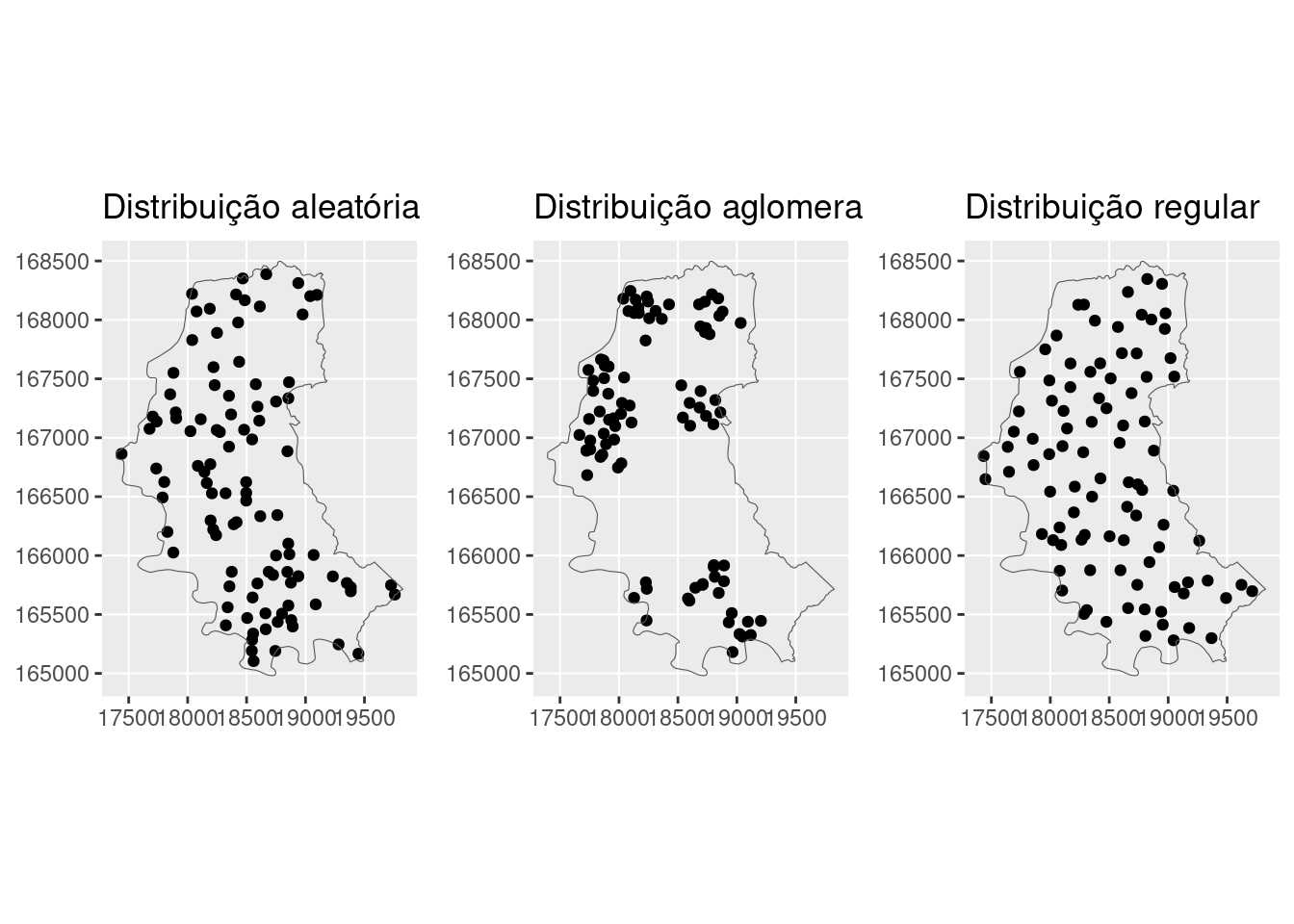
Através da função quadratcount() do pacote spatstat, podemos analisar a contagem de observações por quadrantes, que nos auxilia na análise da distribuição dos pontos. Para isso, precisamos converter nossos objetos para o formato específico ppp (spatial point pattern):
# Convertendo para a class ppp
alea_ppp <- alea_sf2 %>%
as.ppp()
uni_ppp <- uni_sf2 %>%
as.ppp()
clu_ppp <- clu_sf2 %>%
as.ppp()
# Construindo os quadrantes com as respectivas
# contagens
alea_qc <- quadratcount(alea_ppp, nx = 5, ny = 6)
uni_qc <- quadratcount(uni_ppp, nx = 5, ny = 6)
clu_qc <- quadratcount(clu_ppp, nx = 5, ny = 6)
par(mfrow = c(1, 3))
plot(alea_qc, main = "Aleatório")
plot(alea_ppp, add = TRUE, col = "gray")
plot(uni_qc, main = "Regular")
plot(uni_ppp, add = TRUE, col = "gray")
plot(clu_qc, main = "Agregado")
plot(clu_ppp, add = TRUE, col = "gray")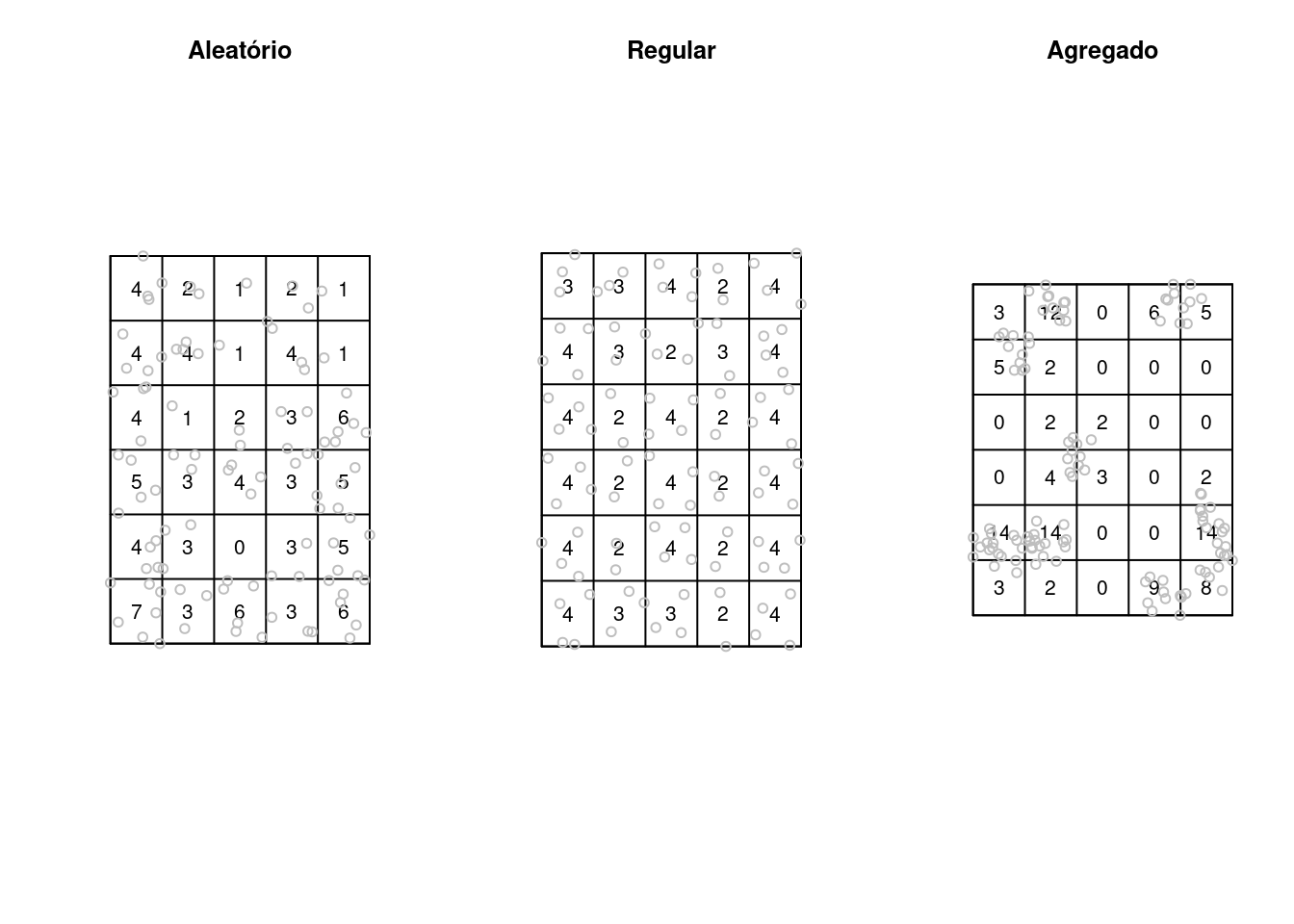
Testando a Completa Aletoriedade Espacial (CSR - complete spatial randomness):
Chi-squared test of CSR using quadrat counts
data:
X2 = 27, df = 29, p-value = 0.9
alternative hypothesis: two.sided
Quadrats: 5 by 6 grid of tiles
Chi-squared test of CSR using quadrat counts
data:
X2 = 170, df = 29, p-value <2e-16
alternative hypothesis: two.sided
Quadrats: 5 by 6 grid of tiles
Chi-squared test of CSR using quadrat counts
data:
X2 = 7.1, df = 29, p-value = 2e-05
alternative hypothesis: two.sided
Quadrats: 5 by 6 grid of tiles7.5 Estimativa de Kernel
Uma análise exploratória de um processo pontual geralmente é iniciada pela estimação da intensidade de ocorrências do processo em toda a região em estudo. Com isso, gera-se uma superfície cujo valor é proporcional à intensidade de eventos por unidade de área.
O estimador Kernel é um interpolador, que possibilita a estimação da intensidade do evento em toda a área, mesmo nas regiões onde o processo não tenha gerado nenhuma ocorrência real.
Essa técnica de alisamento, utiliza janela móvel que, para cada área, estima um peso variável conforme a distância.
O objetivo é de estimar a intensidade do processo pontual \(=\) número de eventos por unidade de área.
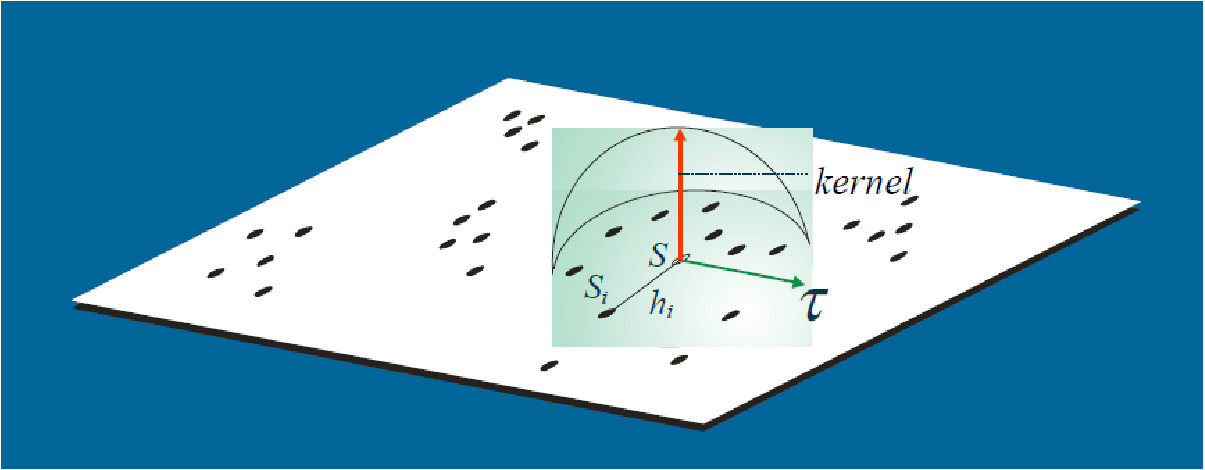
\[\hat{\lambda}(s) = \sum\limits_{i=1}^{n} \dfrac{1}{\tau^2} K \left( \frac{(s - s_i)}{\tau} \right) \nonumber\]
Sendo:
\(\hat{\lambda}(s)\) o valor estimado por área;
A função \(K(\bullet)\) uma FDP, escolhida de forma adequada para construir uma superfície contínua sobre os dados;
O parâmetro \(\tau\) denominado “largura de banda ou faixa” (bandwidth), controla a suavização da superfície gerada;
\(s\) o centro da área, representado por uma localização qualquer na área de estudo;
\(s_i\) as localizações dos eventos observados;
\(n\) o número de pontos (eventos).
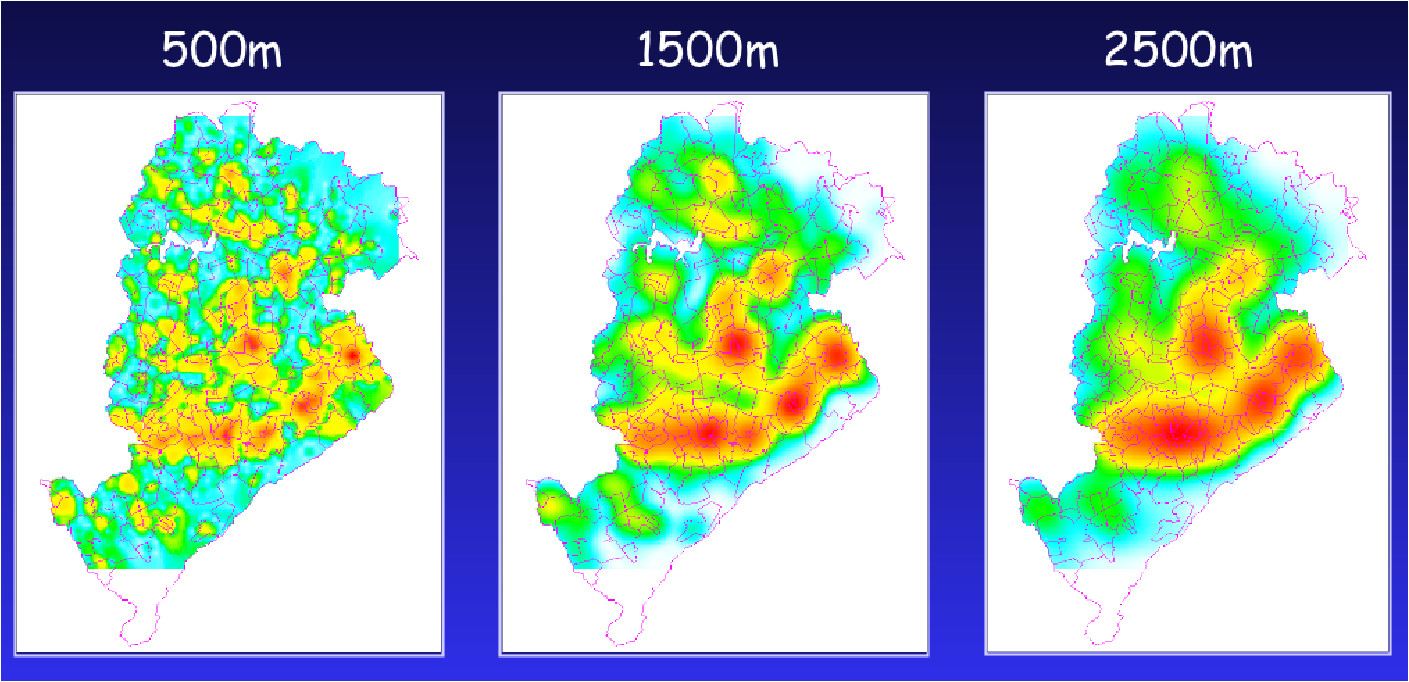
7.5.1 Estimativa de Kernel com correção por bordas
Primeiramente calcula-se o volume sob o Kernel que está de fato dentro da região de estudo.
\[\delta_{\tau}(s) = \int_{R}\dfrac{1}{\tau^2}k \left( \dfrac{(s-u)}{\tau}\right) du\]
Aplicando a correção das bordas, obtém-se um estimador corrigido:
\[\hat{\lambda}(s) = \dfrac{1}{\delta_{\tau}(s)} \sum\limits_{i=1}^{n} \dfrac{1}{\tau^2} K \left( \frac{(s - s_i)}{\tau} \right) \nonumber\]
Exemplo:
alea_den <- density(alea_ppp, sigma = 100)
clu_den <- density(clu_ppp, sigma = 100)
uni_den <- density(uni_ppp, sigma = 100)
par(mfrow = c(1, 3))
plot(alea_den, main = "Aleatório")
plot(alea_ppp, add = T, col = "white", pch = 20)
plot(clu_den, main = "Aglomerado")
plot(clu_ppp, add = T, col = "white", pch = 20)
plot(uni_den, main = "Regular")
plot(uni_ppp, add = T, col = "white", pch = 20)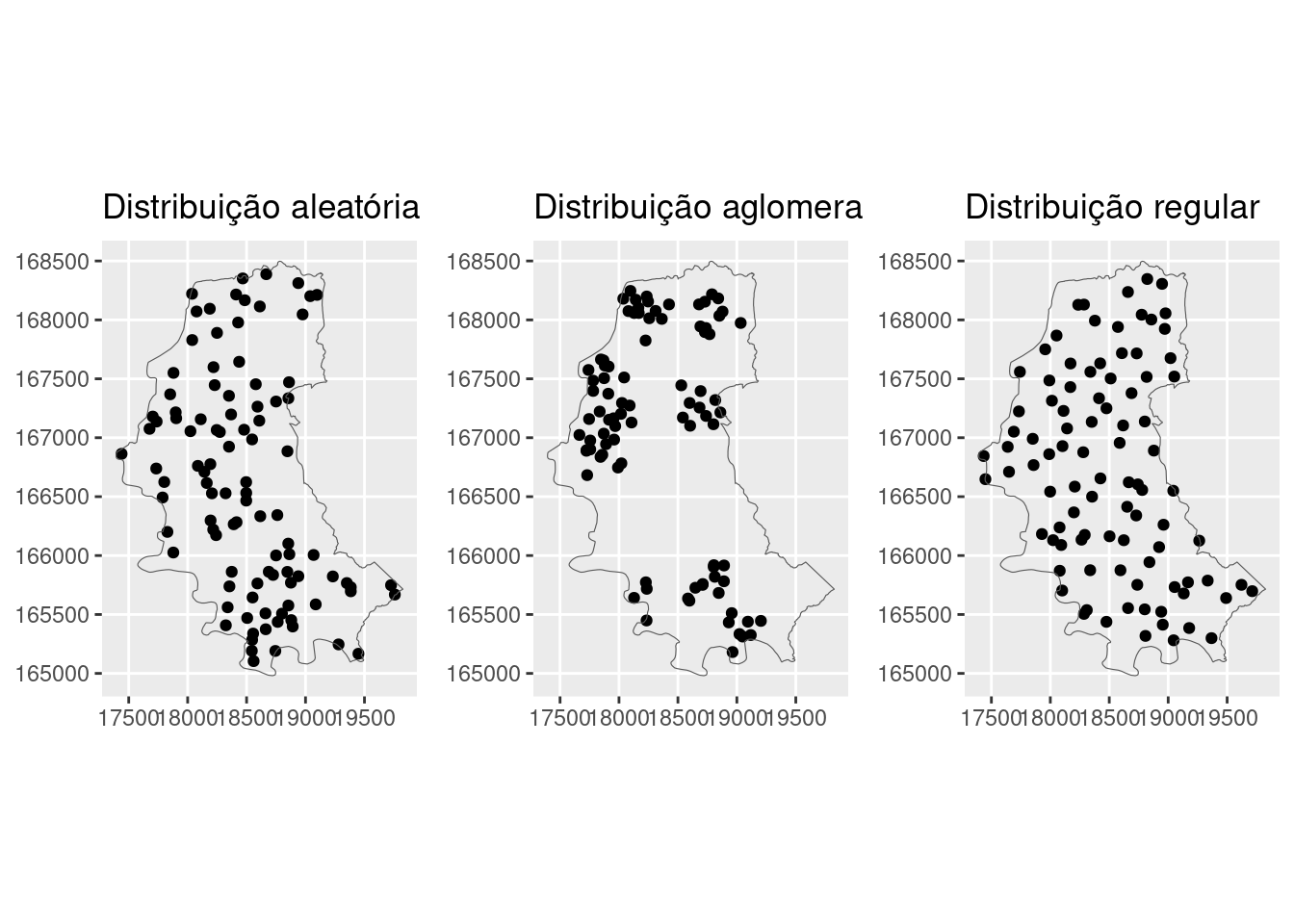
Na própria função density() (veja: ?density.ppp) podemos alterar alguns parâmetros para o kernel, como sua largura de banda (sigma), tipo de Kernel (kernel), correção de bordas (diggle), vetor de pesos (weights), etc.
Alterando a largura de banda de forma exploratória para os dados clusterizados:
clu_den1 <- density(clu_ppp, sigma = 200)
clu_den2 <- density(clu_ppp, sigma = 500)
clu_den3 <- density(clu_ppp, sigma = 1000)
# vamos apresentar agora os gráficos em ggplot
g_den1 <- ggplot(as_tibble(clu_den1), aes(x, y)) +
geom_tile(aes(fill = value)) + geom_point(data = as_tibble(clu_ppp),
color = "white") + scale_fill_viridis_c(option = "B") +
labs(title = "Cluster", subtitle = "Sigma = 200") +
guides(fill = "none") + theme_void()
g_den2 <- ggplot(as_tibble(clu_den2), aes(x, y)) +
geom_tile(aes(fill = value)) + geom_point(data = as_tibble(clu_ppp),
color = "white") + scale_fill_viridis_c(option = "B") +
labs(title = "Cluster", subtitle = "Sigma = 500") +
guides(fill = "none") + theme_void()
g_den3 <- ggplot(as_tibble(clu_den3), aes(x, y)) +
geom_tile(aes(fill = value)) + geom_point(data = as_tibble(clu_ppp),
color = "white") + scale_fill_viridis_c(option = "B") +
labs(title = "Cluster", subtitle = "Sigma = 1000") +
guides(fill = "none") + theme_void()
gridExtra::grid.arrange(g_den1, g_den2, g_den3, ncol = 3)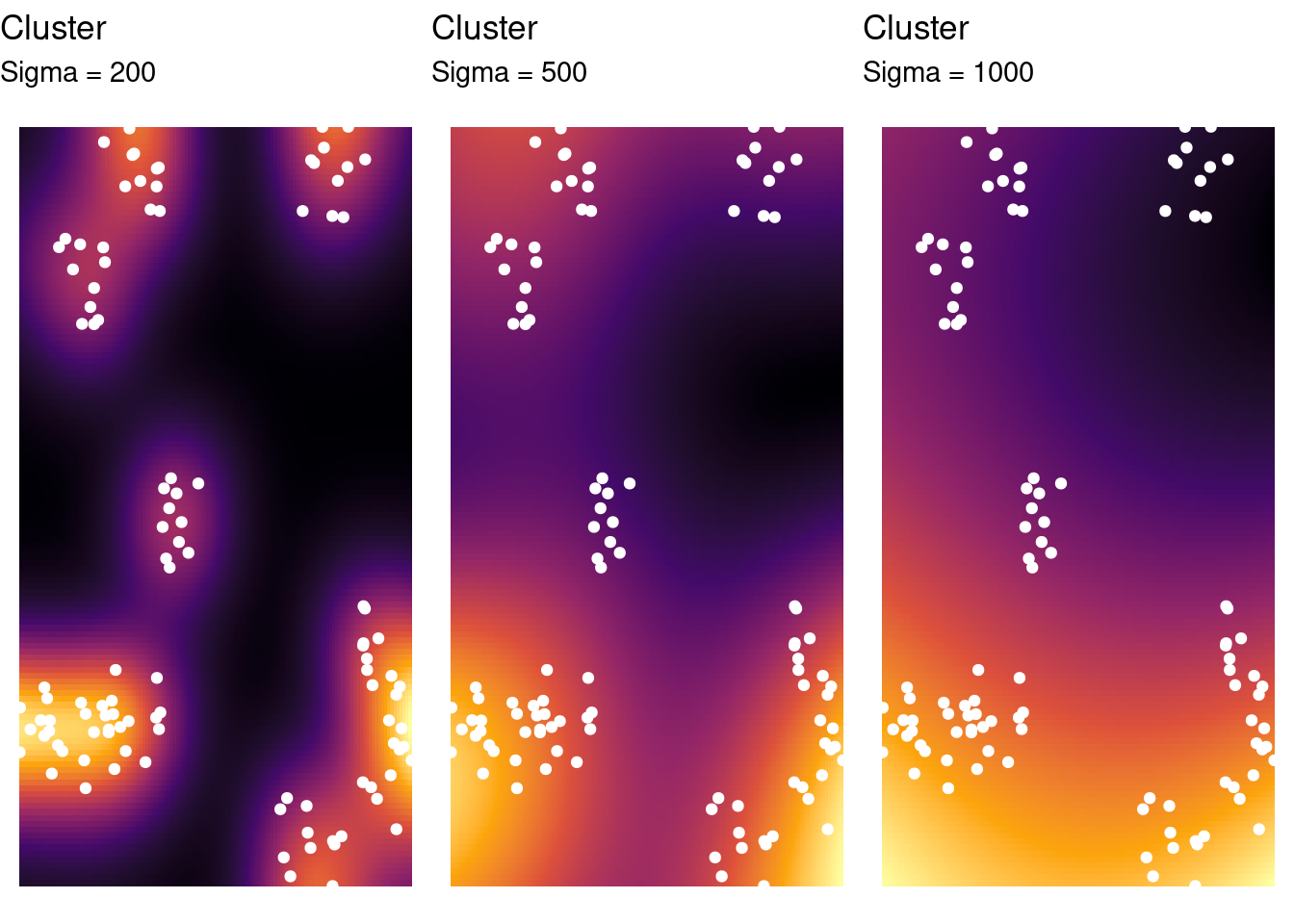
Alterando o tipo de kernel (padrão: "gaussian"):
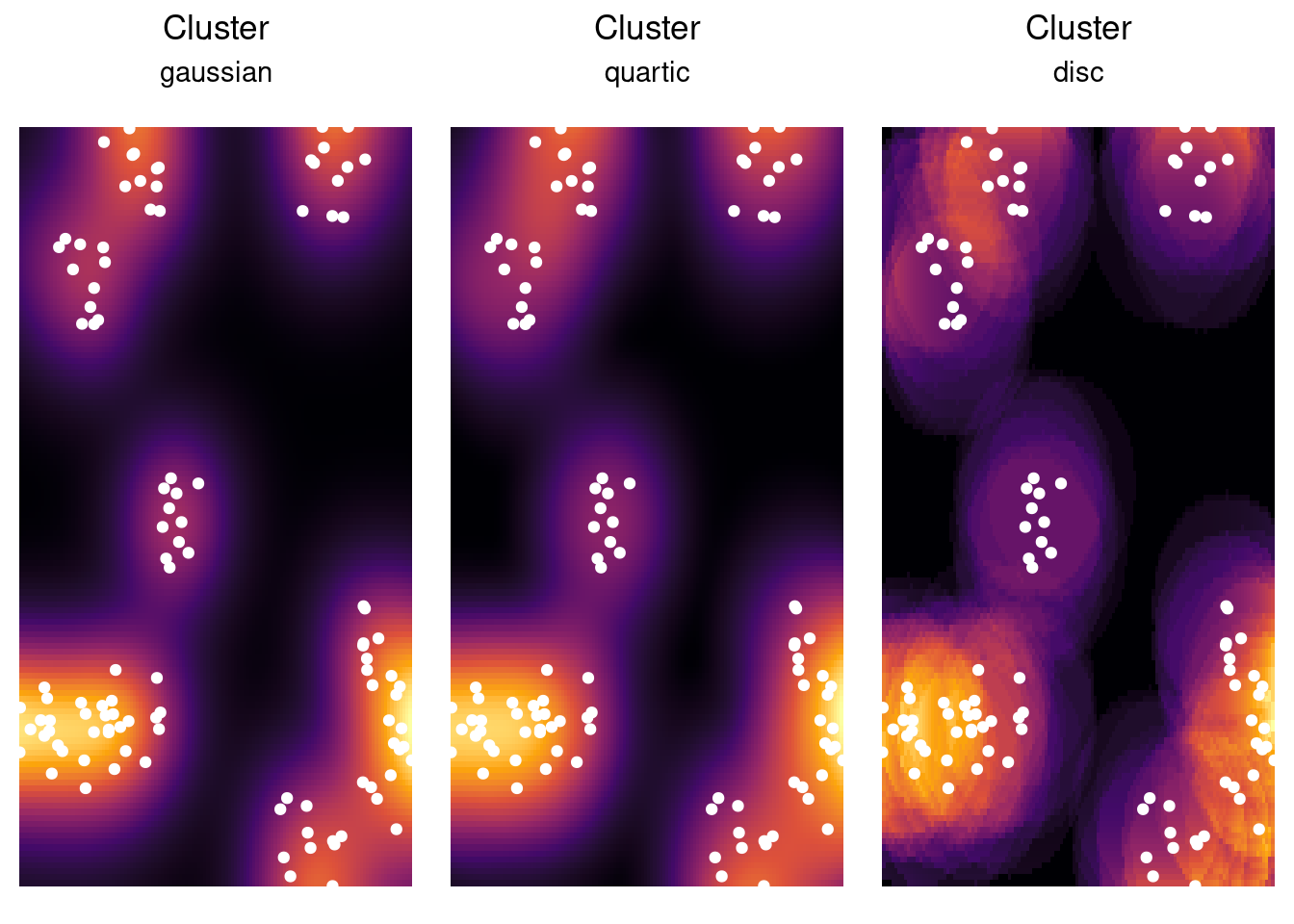
Com correção de bordas:
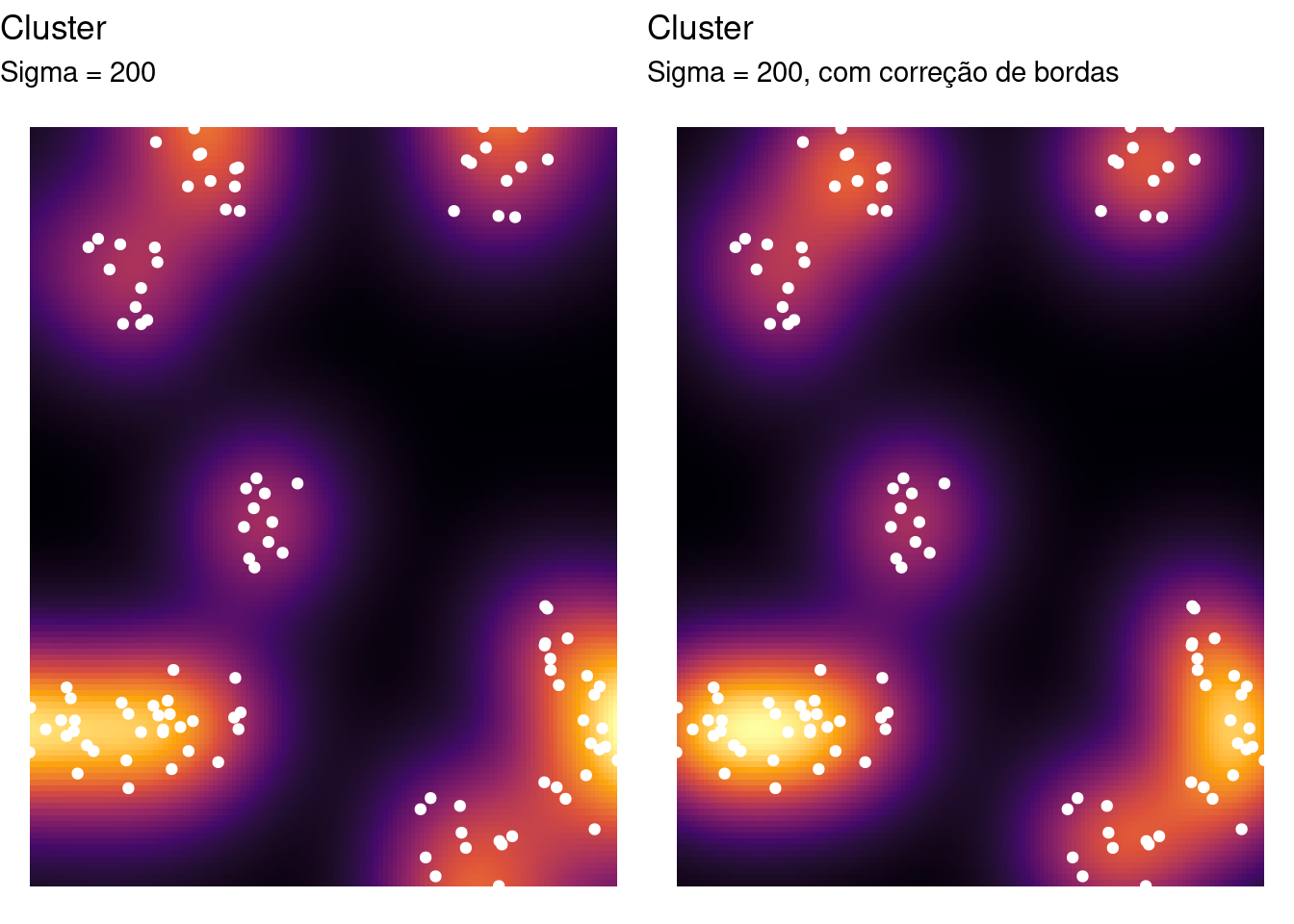
O ggplot possui funções próprias que permitem a visualizar de estimativas de densidade, entre elas o kernel espacial. Nesses casos, o kernel é ajustado na própria função de visualização (stat_density2d).
# vamos inserir linhas de contorno também
g_contour1 <- ggplot(as_tibble(alea_ppp), aes(x, y)) +
stat_density2d_filled(h = c(500, 500)) + stat_density2d(h = c(500,
500), n = 200, contour_var = "count") + geom_point(color = "white") +
labs(title = "Aleatório") + guides(fill = "none") +
theme_void()
g_contour2 <- ggplot(as_tibble(clu_ppp), aes(x, y)) +
stat_density2d_filled(h = c(500, 500)) + stat_density2d(h = c(500,
500), n = 200, contour_var = "count") + geom_point(color = "white") +
labs(title = "Cluster") + guides(fill = "none") +
theme_void()
g_contour3 <- ggplot(as_tibble(uni_ppp), aes(x, y)) +
stat_density2d_filled(h = c(500, 500)) + stat_density2d(h = c(500,
500), n = 200, contour_var = "count") + geom_point(color = "white") +
labs(title = "Regular") + guides(fill = "none") +
theme_void()
gridExtra::grid.arrange(g_contour1, g_contour2, g_contour3,
ncol = 3)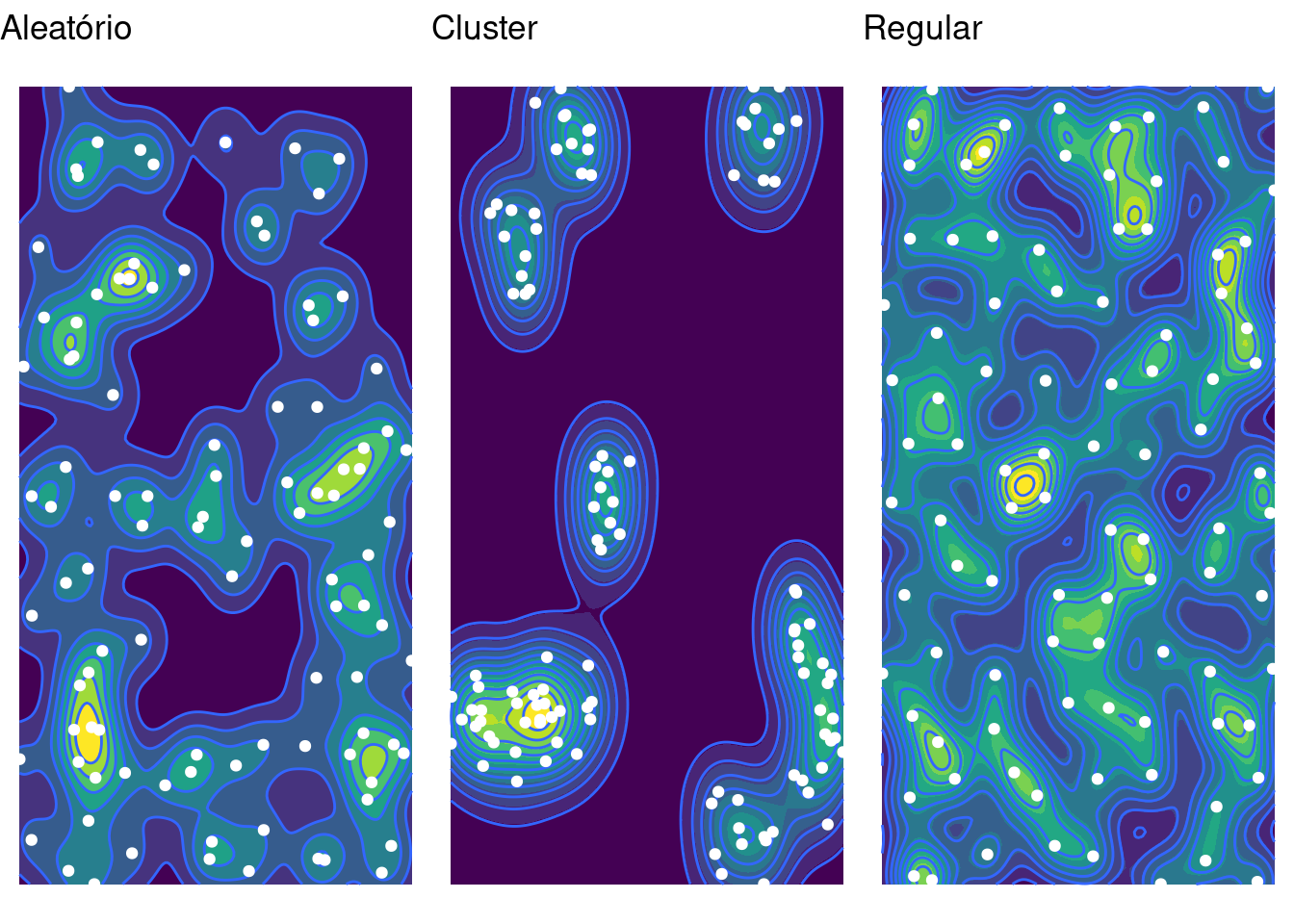
As linhas de contorno também podem ser visualizadas com a função contour() do R base:
par(mfrow = c(1, 3))
plot(alea_den, main = "Aleatório")
contour(alea_den, add = T, col = "white")
plot(clu_den, main = "Aglomerado")
contour(clu_den, add = T, col = "white")
plot(uni_den, main = "Regular")
contour(uni_den, add = T, col = "white")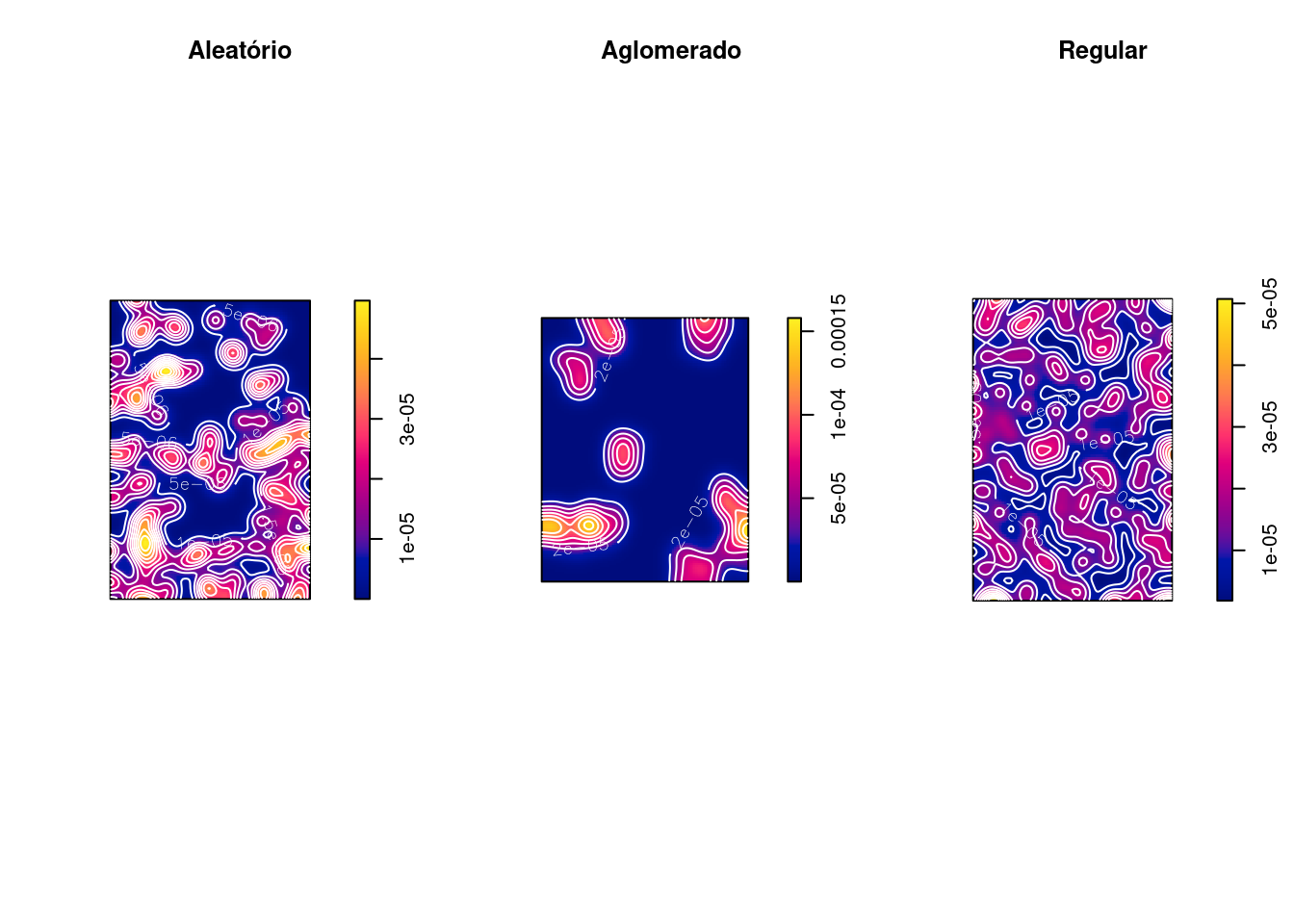
O pacote MASS possui uma função que determina uma largura de banda mais adequada para a estimação do kernel, e é a opção default quando visualizamos um kernel com ggplot(). O pacote splancs também implementa essa funcionalidade utilizando outra metodologia, atravé da função Mse2d().
[1] 1118 1450
g_contour_opt <- ggplot(as_tibble(clu_ppp), aes(x,
y)) + stat_density2d_filled(h = opt_bw) + stat_density2d(h = opt_bw,
n = 200, contour_var = "count") + geom_point(color = "white") +
labs(title = "Cluster", subtitle = "Largura de banda ótima") +
guides(fill = "none") + theme_void()
gridExtra::grid.arrange(g_contour2 + labs(subtitle = "Largura de banda arbitrária (500)"),
g_contour_opt, ncol = 2)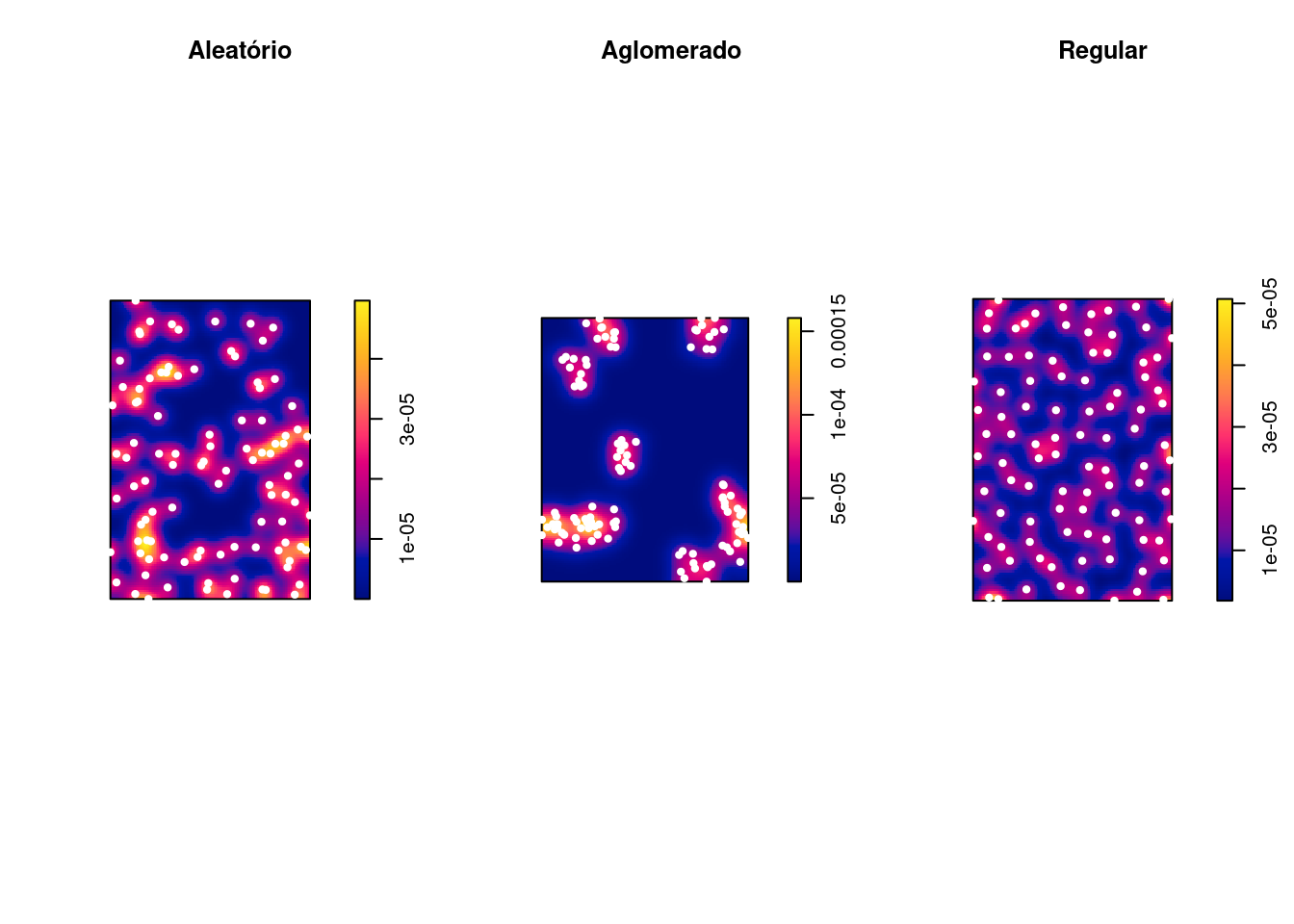
7.6 Kernel por atributo
O alisamento utilizando o Kernel permite estimar a densidade de eventos por unidade de área. É possível, também, se usar uma covariável (atributo).
Como exemplo, pode-se estimar a “população por unidade de área”, e até mesmo fazer a razão entre dois Kernels obtendo uma estimativa alisada de “eventos por população”.
\[\hat{\lambda}(s) = \sum\limits_{i=1}^{n} \dfrac{1}{\tau^2} K \left( \frac{(s - s'_j)}{\tau} \right) y_i \nonumber\]
Sendo:
\(\lambda'\) : Estimativa do atributo para unidade de área
\(\tau\) : Largura de banda
\(y_i\) : valor do atributo em cada ponto
Pode-se atribuir ao centróide do setor censitário ou ao centro populacional o número de habitantes de toda a área (atributo), por exemplo.
7.7 Razão de Kernel
Vamos criar uma “taxa suavizada” a partir da divisão dos alisamentos dos eventos por unidade de área dividido pelo alisamento da população por unidade de área.
\[\hat{\lambda}(s) = \dfrac{\sum\limits_{i=1}^{n} \dfrac{1}{\tau^2} K \left( \frac{(s - s_i)}{\tau} \right)}{\sum\limits_{i=1}^{n} \dfrac{1}{\tau^2} K \left( \frac{(s - s'_j)}{\tau} \right) y_i} \nonumber\]
Pode-se usar diferentes larguras de banda (em geral maior no denominador para estabilizar mais).
Pode-se usar outro evento pontual como “estimador da população a risco”.
Exemplo: Comparando 2 eventos: casos clusterizados (casos) vs casos aleatórios (controle):
alea_den1 <- density(alea_ppp, sigma = 200)
clu_alea_ratio <- clu_den1/alea_den1
par(mfrow = c(1, 3))
plot(clu_den1, main = "Cluster")
plot(clu_ppp, add = T, col = "white", pch = 20)
plot(alea_den1, main = "Aleatório")
plot(alea_ppp, add = T, col = "white", pch = 20)
plot(clu_alea_ratio, main = "Razão")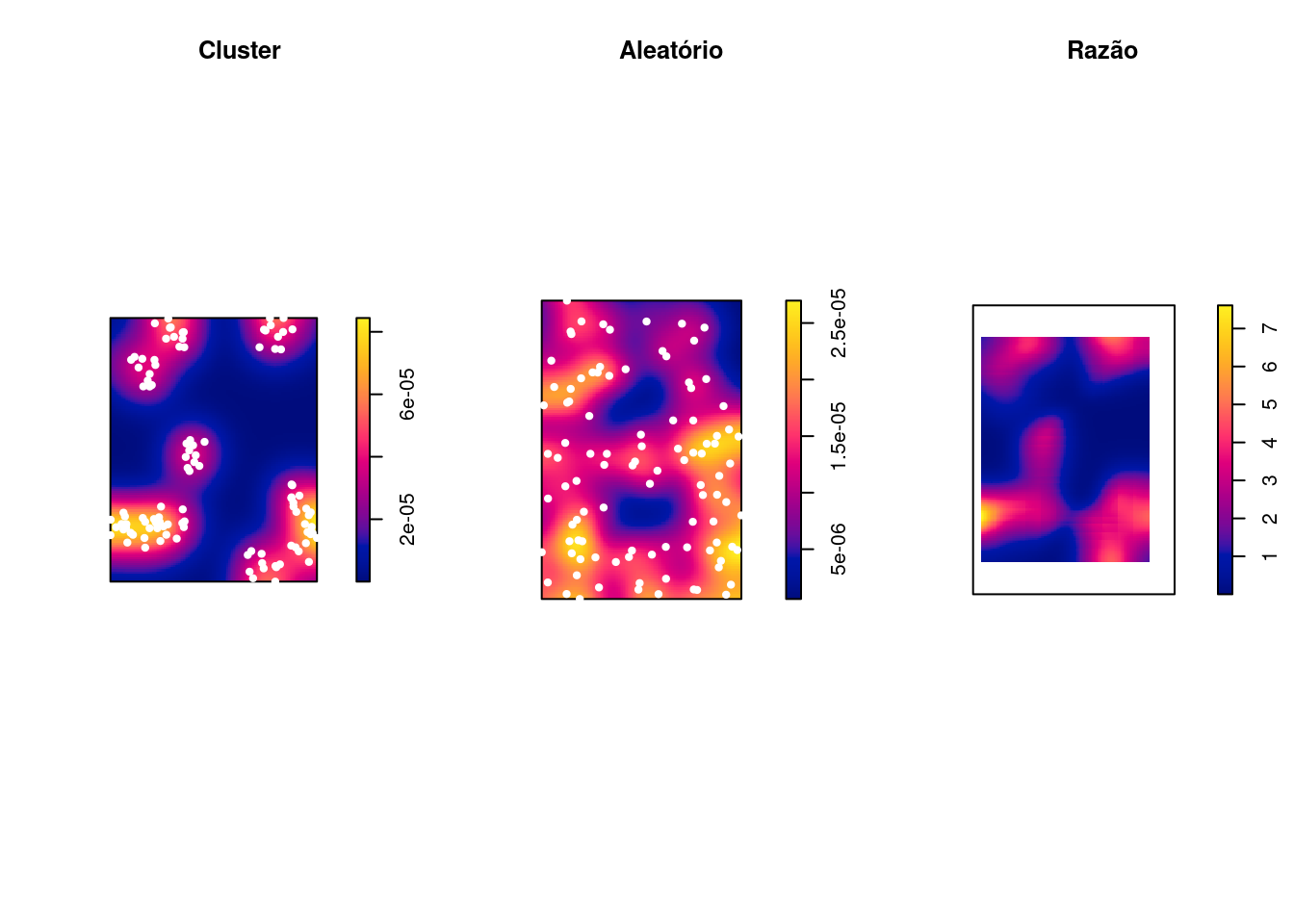
Exemplo: Verificando o padrão espacial de segunda ordem dos casos de homicídos em POA/RS:
# Além de transformar os pontos para ppp, vamos
# especificar uma janela para delimitar a área de
# estimação para o kernel:
homic_ppp <- homic_sf2 %>%
as.ppp()
Window(homic_ppp) <- as.owin(poa_sf)
homic_den1 <- density(homic_ppp, sigma = 200, diggle = TRUE)
g_homic_den1 <- ggplot(as_tibble(homic_den1), aes(x,
y)) + geom_tile(aes(fill = value)) + geom_point(data = as_tibble(homic_ppp),
color = "white", shape = 1, size = 0.5) + geom_sf(data = poa_sf,
aes(geometry = geometry), fill = "transparent",
inherit.aes = FALSE) + scale_fill_viridis_c(option = "B") +
labs(title = "Homicídios", subtitle = "sigma = 200") +
guides(fill = "none") + theme_void()
suic_ppp <- suic_sf %>%
as.ppp()
Window(suic_ppp) <- as.owin(poa_sf)
suic_den1 <- density(suic_ppp, sigma = 200, diggle = TRUE)
g_suic_den1 <- ggplot(as_tibble(suic_den1), aes(x,
y)) + geom_tile(aes(fill = value)) + geom_point(data = as_tibble(suic_ppp),
color = "white", shape = 1, size = 0.5) + geom_sf(data = poa_sf,
aes(geometry = geometry), fill = "transparent",
inherit.aes = FALSE) + scale_fill_viridis_c(option = "B") +
labs(title = "Suicídios", subtitle = "sigma = 200") +
guides(fill = "none") + theme_void()
acid_ppp <- acid_sf %>%
as.ppp()
Window(acid_ppp) <- as.owin(poa_sf)
acid_den1 <- density(acid_ppp, sigma = 200, diggle = TRUE)
g_acid_den1 <- ggplot(as_tibble(acid_den1), aes(x,
y)) + geom_tile(aes(fill = value)) + geom_point(data = as_tibble(acid_ppp),
color = "white", shape = 1, size = 0.5) + geom_sf(data = poa_sf,
aes(geometry = geometry), fill = "transparent",
inherit.aes = FALSE) + scale_fill_viridis_c(option = "B") +
labs(title = "Acidentes", subtitle = "sigma = 200") +
guides(fill = "none") + theme_void()
gridExtra::grid.arrange(g_homic_den1, g_suic_den1,
g_acid_den1, ncol = 3)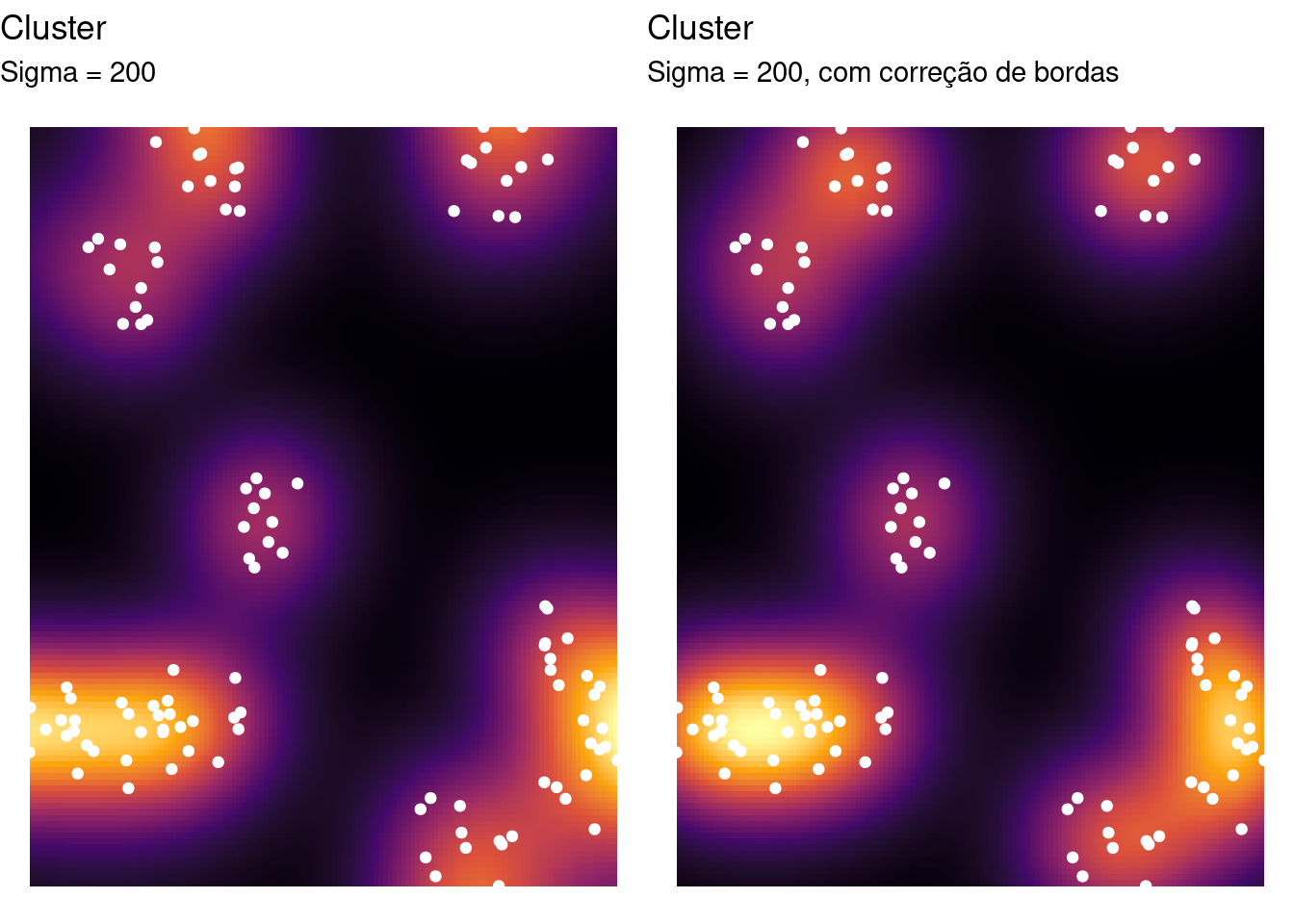
homic_den2 <- density(homic_ppp, sigma = 100, diggle = TRUE)
g_homic_den2 <- ggplot(as_tibble(homic_den2), aes(x,
y)) + geom_tile(aes(fill = value)) + geom_point(data = as_tibble(homic_ppp),
color = "white", shape = 1, size = 0.5) + geom_sf(data = poa_sf,
aes(geometry = geometry), fill = "transparent",
inherit.aes = FALSE) + scale_fill_viridis_c(option = "B") +
labs(title = "Homicídios", subtitle = "sigma = 100") +
guides(fill = "none") + theme_void()
homic_den3 <- density(homic_ppp, sigma = 500, diggle = TRUE)
g_homic_den3 <- ggplot(as_tibble(homic_den3), aes(x,
y)) + geom_tile(aes(fill = value)) + geom_point(data = as_tibble(homic_ppp),
color = "white", shape = 1, size = 0.5) + geom_sf(data = poa_sf,
aes(geometry = geometry), fill = "transparent",
inherit.aes = FALSE) + scale_fill_viridis_c(option = "B") +
labs(title = "Homicídios", subtitle = "sigma = 500") +
guides(fill = "none") + theme_void()
gridExtra::grid.arrange(g_homic_den2, g_homic_den1,
g_homic_den3, ncol = 3)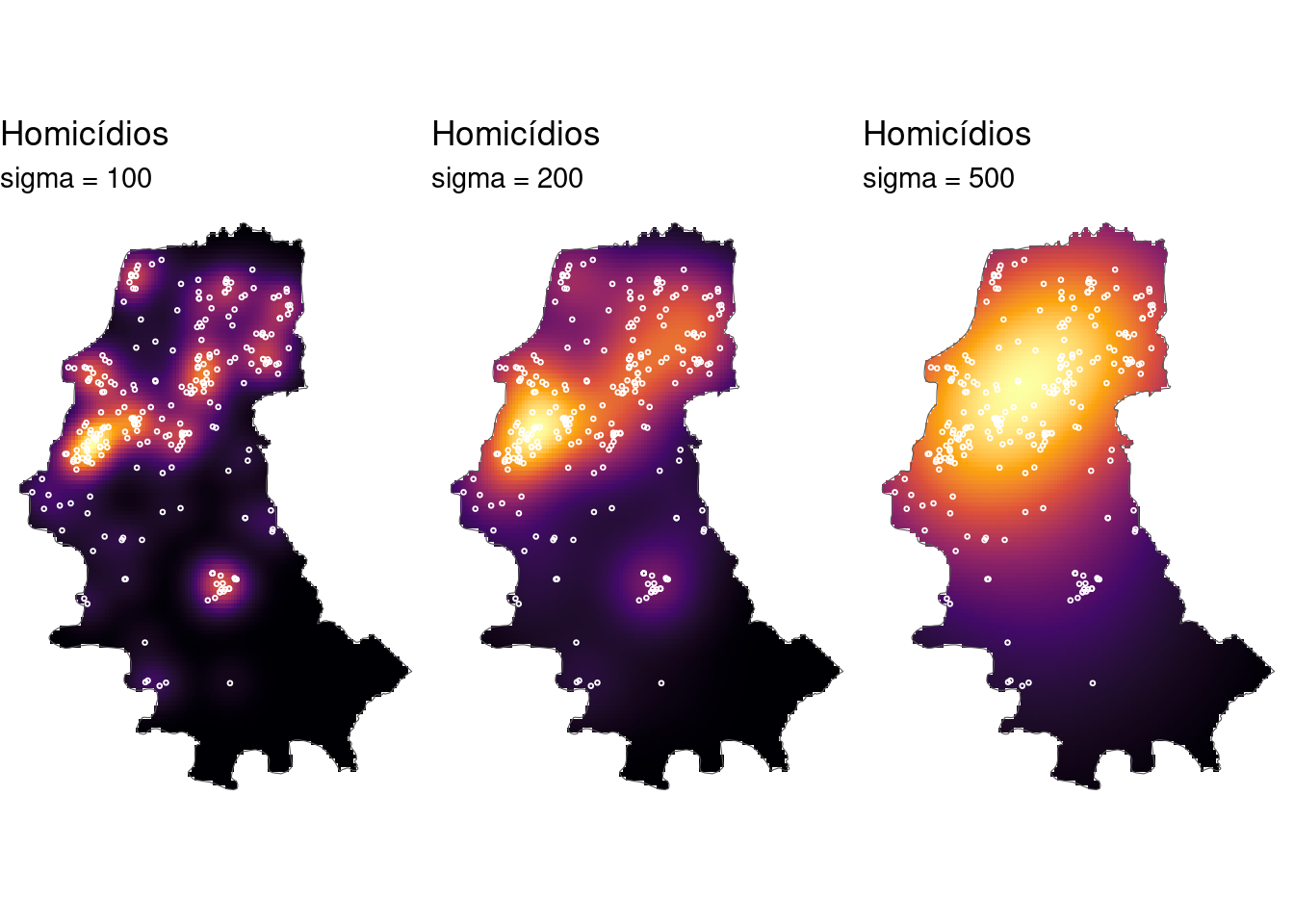
Exemplo: Fazendo a razão de kernel entre as causas de homicídio e suicídios de Porto alegre/RS:
suic_homic_ratio <- suic_den1/homic_den1
g_suic_homic <- ggplot(as_tibble(suic_homic_ratio),
aes(x, y)) + geom_tile(aes(fill = value)) + geom_sf(data = poa_sf,
aes(geometry = geometry), fill = "transparent",
inherit.aes = FALSE) + scale_fill_viridis_c(option = "B") +
labs(title = "Razão", subtitle = "Suicídios/Homicídios") +
guides(fill = "none") + theme_void()
gridExtra::grid.arrange(g_homic_den1, g_suic_den1,
g_suic_homic, ncol = 3)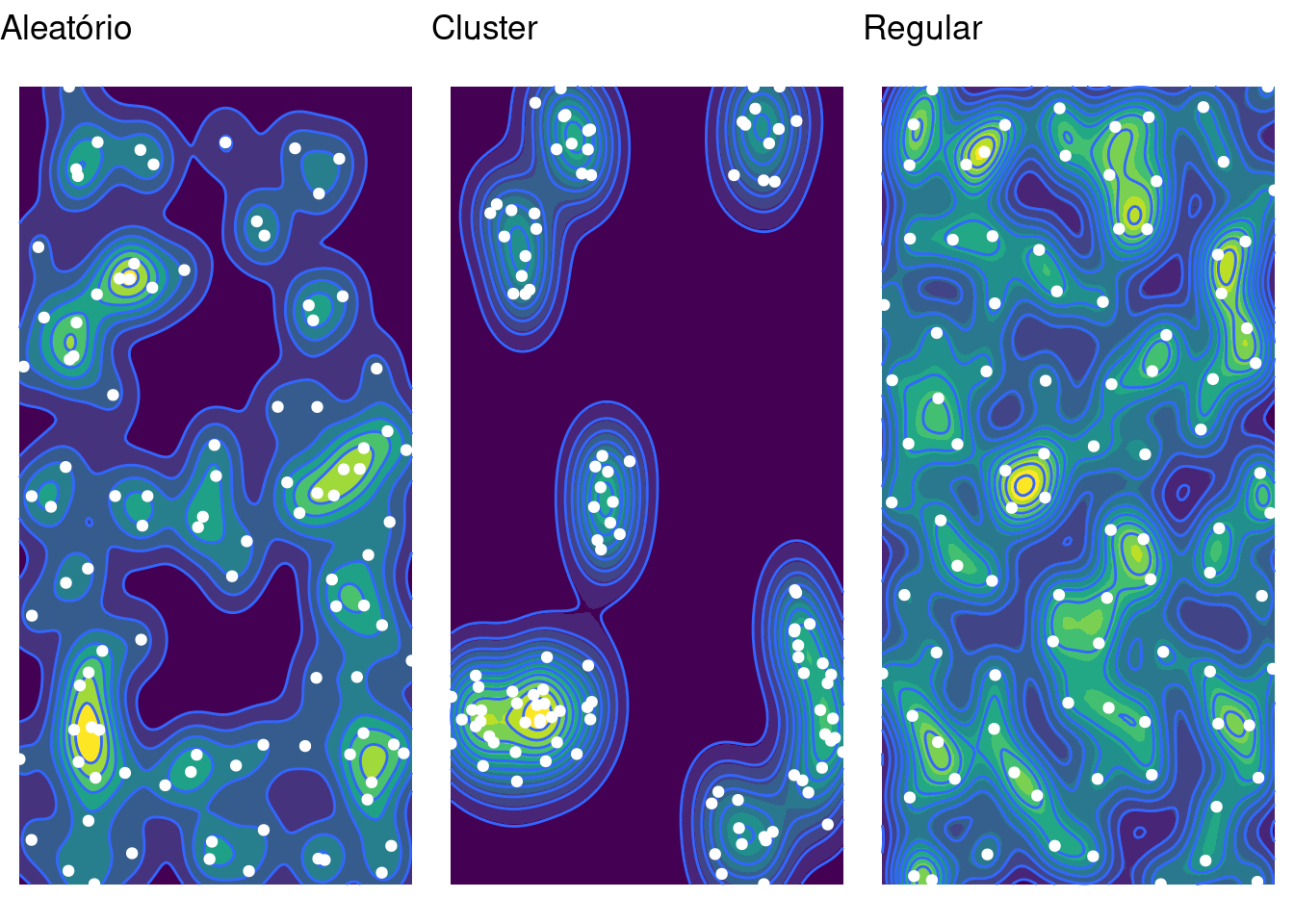
7.8 Análise preliminar de um processo pontual
7.8.1 Função F e G - Distância do vizinho mais próximo
Kernel e quadrat permitem explorar a variação da média do processo na região de estudo (propriedade de primeira ordem).
Para investigar a propriedade de segunda ordem é necessário observar as distâncias entre os eventos.
O método do vizinho mais próximo estima a função de distribuição cumulativa baseado nas distâncias entre eventos ou pontos em uma região de análise.
Dois tipos de distâncias: evento-evento (W) e ponto aleatório-evento (X)
\[\hat{F}(x) = \dfrac{\#(x_i \leq x)}{m}\]
\[\hat{G}(w) = \dfrac{\#(w_i \leq w)}{n}\]
- Sabendo que:
W - evento-evento
X - ponto-evento
# - contagem de pontos onde a condição acontece
n - total de eventos
m - total de pontos aleatórios
Podemos dizer que ambas as funções podem ser representadas pelo número de (\(x_i\)) ou (\(w_i\)) cuja distância é menor ou igual ao evento (\(x\)) ou ponto (\(w\)), dividido pelo total de pontos (\(m\)) ou total de eventos (\(n\)) na região.
O resultado desta função empírica é o histograma das distâncias para o vizinho mais próximo - cada classe do histograma é uma contagem de eventos que ocorrem até aquela distância.
A plotagem dos resultados desta função de distribuição cumulativa empírica pode ser usada como um método exploratório para verificar se existe evidência de interação entre os eventos. Se esta plotagem apresentar um crescimento rápido para pequenos valores de distância, esta situação aponta para interação entre os eventos caracterizando agrupamentos nestas escalas. Se esta plotagem apresentar valores pequenos no seu início, e só crescer rapidamente para valores maiores de distância, esta situação aponta para uma distribuição mais regular.
par(mfrow = c(1, 3))
plot(envelope(Y = alea_ppp, fun = Gest, nsim = 99),
main = "Aleatório")
plot(envelope(Y = uni_ppp, fun = Gest, nsim = 99),
main = "Regular")
plot(envelope(Y = clu_ppp, fun = Gest, nsim = 99),
main = "Agregado")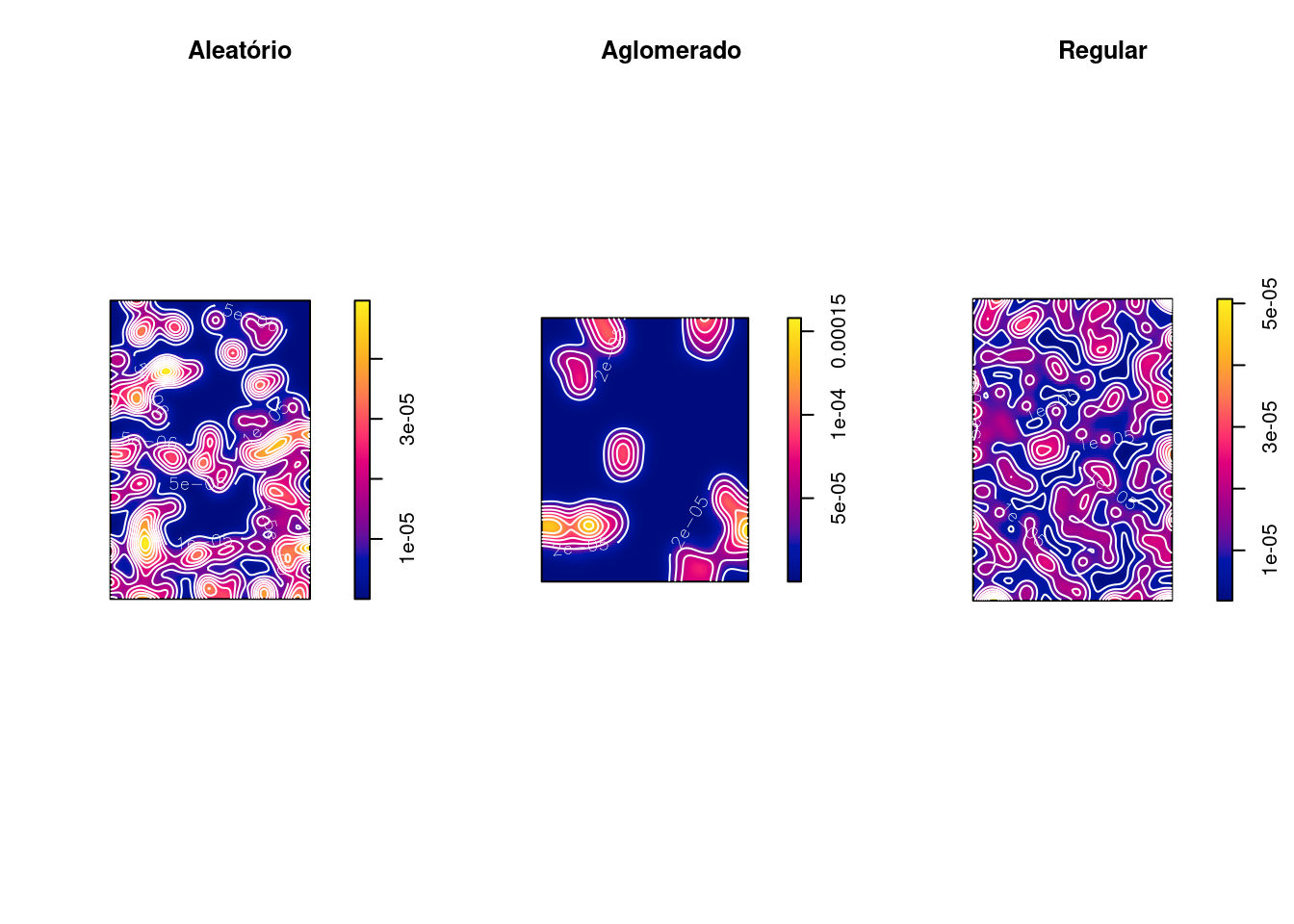 Embora o método do vizinho mais próximo forneça uma indicação inicial da distribuição espacial, ele considera apenas escalas pequenas. Para se ter informação mais efetiva para o padrão espacial em escalas maiores, o melhor método a ser utilizado é o da função \(K\).
Embora o método do vizinho mais próximo forneça uma indicação inicial da distribuição espacial, ele considera apenas escalas pequenas. Para se ter informação mais efetiva para o padrão espacial em escalas maiores, o melhor método a ser utilizado é o da função \(K\).
7.9 Propriedade de segunda ordem
7.9.1 Função K de Ripley (ou apenas função K)
- A função \(K\) permite analisar as propriedades de segunda ordem de um processo isotrópico.
\[\lambda K(h) = E(\#eventos)\]
Sendo:
\(#eventos\) - o número de eventos esperados até distância \(h\)
\(\lambda\) - a intensidade ou número médio de eventos por unidade de área
eventos contidos a uma distância h de um evento arbitrário\()\)
- A função \(K(h)\) é, para cada distância \(h\), o somatório do total de pares cuja distância é menor de que \(h\), vezes o inverso do total de pares ordenados existente na região \(R\).
\[K(h) = \dfrac{1}{\lambda^{2} R}\sum \sum\limits_{i \neq j} I_{h} (d_{ij})\]
Supondo:
\[I_{h}(d_{ij}) = \begin{cases} 1 \ \ se\ \ d_{ij} \leq h \\ 0 \ \ se\ \ d_{ij} > h \end{cases}\]
\(I_{h}(d_{ij})\) é uma função indicadora
Esta função também necessita de correção do efeito de borda;
A função K de Ripley conta quantos pontos há em círculos em torno de uma planta focal;
Os círculos começam com um raio pequeno e vão até um raio que inclui toda a área de estudo;
Faz-se uma média do número de pontos nas classes de distâncias em torno de todas as plantas focais da população.
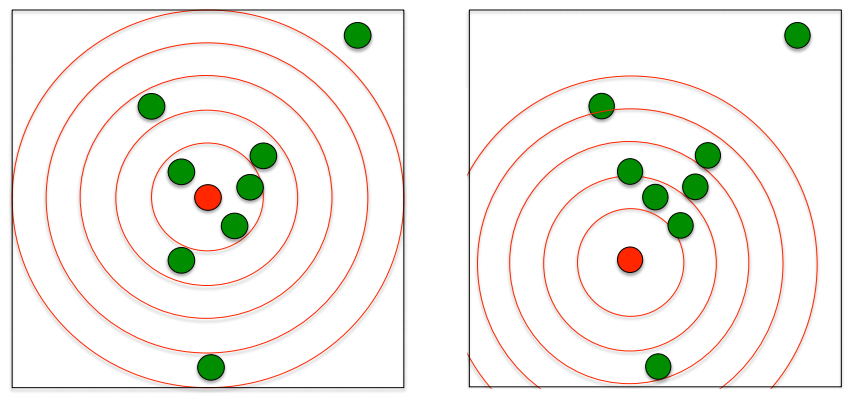
- A distribuição é cumulativa e representa o no esperado de vizinhos em um círculo de raio r centrado em uma planta arbitrária dividido pela intensidade \(\lambda\) do padrão dos pontos na área de estudo;
Possíveis resultados:
quando o processo é completamente aleatório, a curva se desvia relativamente pouco de \(\pi r²\). A curva \(K\) permanece perto de o valor de referência \(\pi r²\);
no caso de um processo regular, obtemos: \(\hat{K}(r) < K_{pois}(r)\) porque se os pontos forem repulsivos, eles têm menos vizinhos em média em um raio \(r\) do que teriam baseado no pressuposto de uma distribuição aleatória de pontos. Graficamente, a curva K reflete isso repulsão: vemos que no gráfico à abaixo, no processo regular, a curva K está localizada abaixo da referência valor (\(\pi r²\));
no caso de um processo agregado, há em média mais pontos em um raio \(r\) ao redor os pontos do que o número esperado sob uma distribuição aleatória: consequentemente, os pontos atraem um ao outro e \(\hat{K}(r) > K_{pois}(r)\). Graficamente, a curva \(K\) está neste momento localizada acima da valor de referência para todas as áreas de estudo.
par(mfrow = c(1, 3))
plot(envelope(Y = alea_ppp, fun = Kest, nsim = 99),
main = "Aleatório")
plot(envelope(Y = uni_ppp, fun = Kest, nsim = 99),
main = "Regular")
plot(envelope(Y = clu_ppp, fun = Kest, nsim = 99),
main = "Agregado")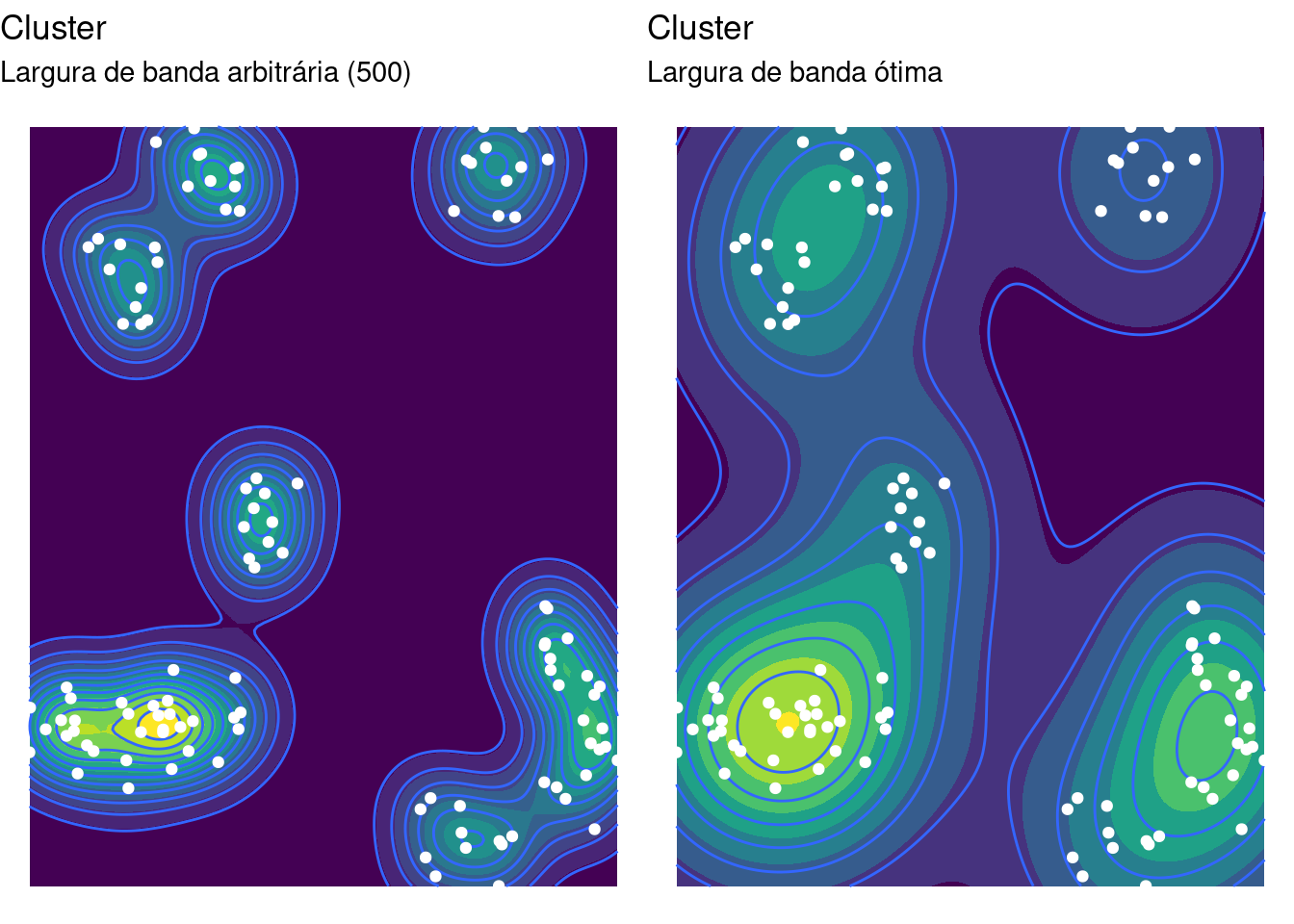
7.9.2 Função L
- A função \(K(h)\) tem uma distribuição teórica sob condições de aleatoriedade, quando a probabilidade de ocorrência de um evento em qualquer ponto de R é independente da ocorrência de outros eventos e igual em toda a superfície.
Neste caso, o número de eventos a uma distância \(h\) será \(\pi \lambda h^2\):
\[ K(h) = \pi h^2\]
No caso de distribuição regular, \(K(h)\) será menor que \(\pi h^2\).
Distribuição em cluster, K(h) será maior que \(\pi h^2\).
A função \(L(h)\) permite comparar a função \(K(h)\) e \(\pi h^2\)
\[L(h) = \sqrt {\dfrac{K(h)}{\pi}}\]
- Picos positivos indicam atração espacial - cluster
- Vales negativos - repulsão espacial ou regularidadepar(mfrow = c(1, 3))
plot(envelope(Y = alea_ppp, fun = Lest, nsim = 99),
main = "Aleatório")
plot(envelope(Y = uni_ppp, fun = Lest, nsim = 99),
main = "Regular")
plot(envelope(Y = clu_ppp, fun = Lest, nsim = 99),
main = "Agregado")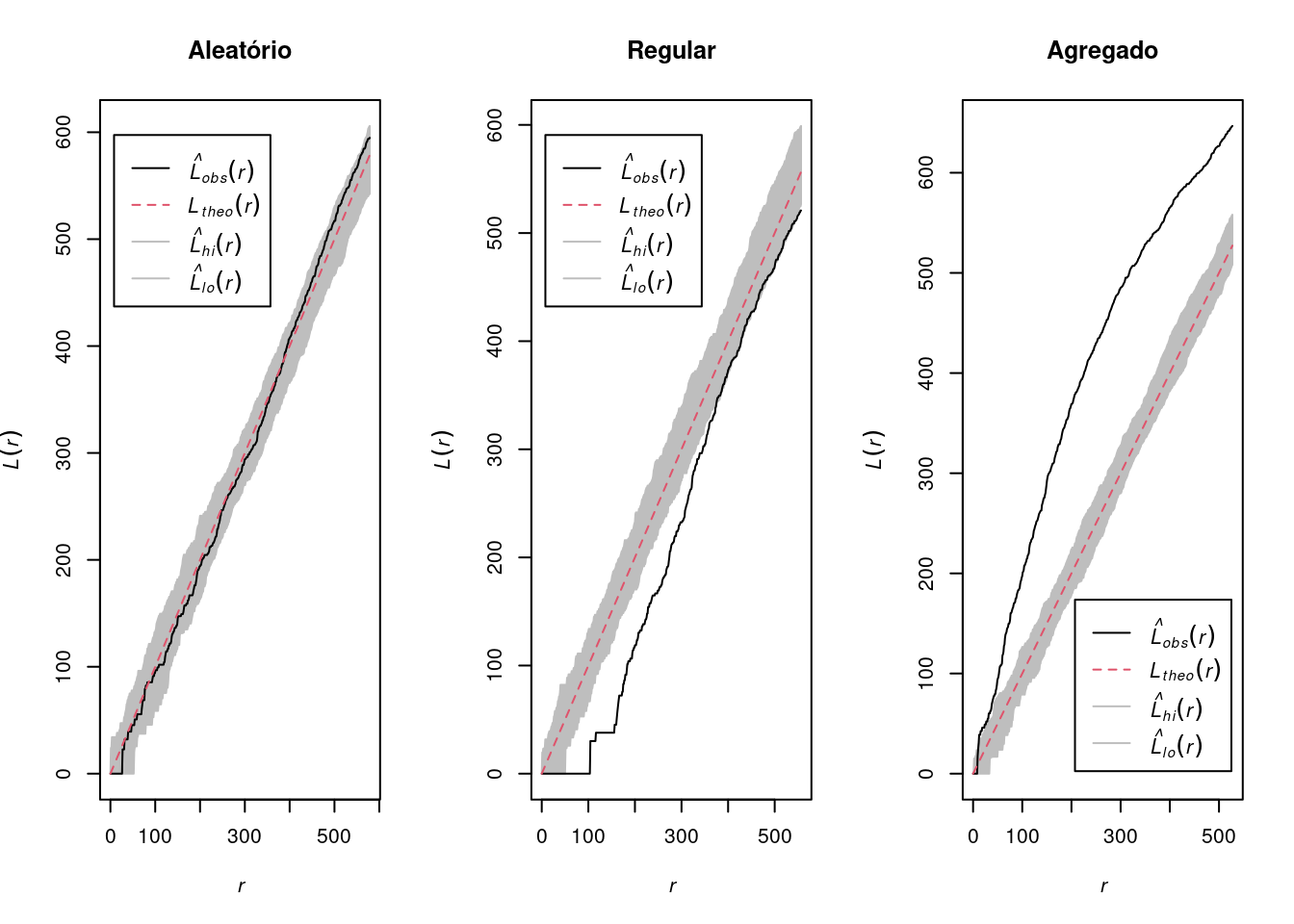
Exemplo: Verificando o padrão espacial de segunda ordem dos casos de homicíodos em POA/RS:
# vamos usar só a geometria para não incluir
# aquela variável 'dentro'
homic_ppp2 <- as.ppp(homic_sf2$geometry)
plot(homic_ppp2, pch = 19, cex = 0.5)
polymap(contorno.poa, add = T)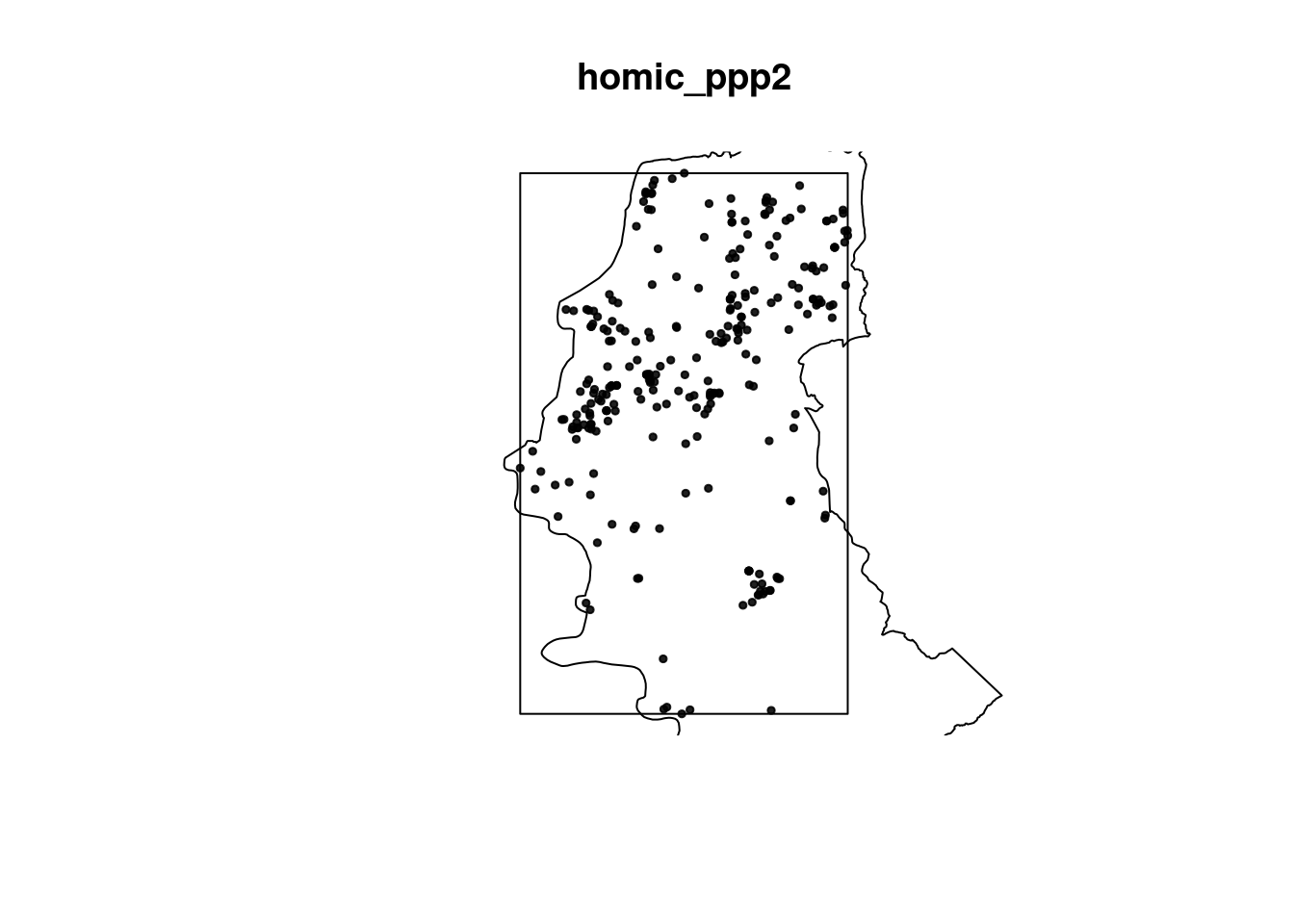
Note que o restangulo envolvente (bbox) foi feito com a coordenadas dos dados, não do contorno de POA.
Grafícos das Funções K , G e L dos homicíodos em POA/RS:
par(mfrow = c(1, 3))
plot(envelope(Y = homic_ppp2, fun = Kest, nsim = 99),
main = "Funcao K")
plot(envelope(Y = homic_ppp2, fun = Gest, nsim = 99),
main = "Funcao G")
plot(envelope(Y = homic_ppp2, fun = Lest, nsim = 99),
main = "Funcao L")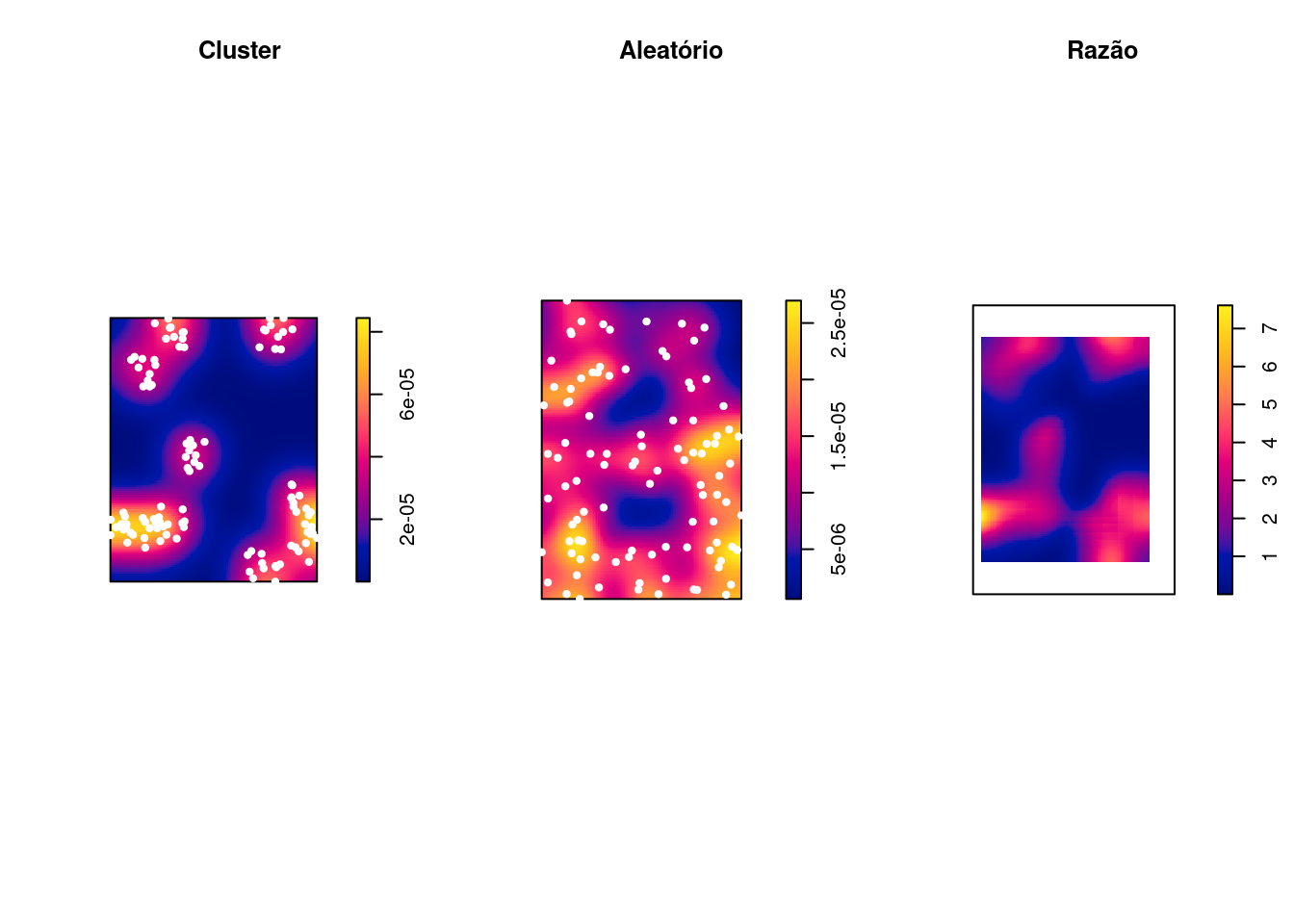
Comente esses resultados!
7.10 Detecção de cluster
Definição (Knox): grupo de ocorrências geograficamente limitado em tamanho e concentração tais que seja improvável ocorrer por mero acaso.
São causas de cluster: fonte comum, contagiosidade.
Clusters são em geral espaço-temporais.
É importante considerar:
Demais fatores de risco – sexo, idade;
Residência X outros locais;
Latência.
Dois tipos básicos de testes:
Focados: testa-se a hipótese de excesso de casos ao redor de fonte suspeita, identificada antes de observar os dados;
Genéricos: busca identificar áreas quentes, sem especificar quais e quantas.
Hipóteses dos testes:
\(H_0\): É ausência de cluster: completa aleatoriedade espacial (CSR).
CSR
Sendo:
\(n\) são subdivisões da região do estudo,
\(y_i\) número de casos observados e \(E_i\) esperados,
\(\lambda\) eventos por unidade de área (e tempo).
Alternativas:
Focados - \(\lambda\) varia com a distância da fonte
Genéricos - existe regiões onde \(\lambda\) é mais elevado
7.10.1 Testes Genéricos de Cluster
Knox: Testa um número acima do esperado de pares de casos excessivamente próximos (segundo critério pré-estabelecido) no espaço e no tempo.
Mantel: Distância no tempo e distância no espaço, se \(x\) for 1 e \(y\) for 1, equivale ao teste de Knox.
\[\sum \sum\limits_{i \neq j} x_{ij} y_{ij} \]
- Cuzick-Edwards: Caso-controle onde a coincidência de casos vizinhos aumenta o peso, e a junção controle-controle ou caso-controle tem peso zero; este teste permite considerar a variação populacional.
7.10.2 Fonte Específica
Cluster ao redor de um ponto ou uma linha.
Compara-se a ocorrência de no excessivo de “casos” em relação à população a partir de uma função de decaimento em relação à possível fonte.
\[\lambda (s) = \rho \lambda'(s)f(h;\theta)\]
\[f(h;\theta) = 1 + \theta_1 e^{\theta_{2}h^2}\]
Sendo:
\(\theta(s)\) - estimativa do evento p/ unidade de área
\(\rho\) - parâmetro que indica a razão entre “casos” e “controles”
\(\lambda'(s)\) - estimativa população p/ unidade de área
\(f\) - função da distância para a fonte
\(θ\) - parâmetros a estimar que descrevem como a incidência varia em torno da fonte
7.11 Exercícios Propostos
Testar a CSR e explorar os Kernels para as causas de suicídio e acidentes de carro em POA/RS.
Fazer a razão de kernel entre suicídios e acidentes de carro em POA/RS.
Inspecionar o processo pontual de segunta ordem, utilizando as funções K, G e L para as causas de suicídio e acidentes de carro em POA/RS.
7.12 Bibliografia sugerida
ASSUNÇÃO, Renato M. Estatística espacial com aplicações em epidemiologia, economia e sociologia. São Carlos: Associação Brasileira de Estatística, v. 131, 2001.
DIGGLE, Peter J. et al. Statistical analysis of spatial point patterns. Academic press, 1983.
GATRELL, Anthony C. et al. Spatial point pattern analysis and its application in geographical epidemiology. Transactions of the Institute of British geographers, p. 256-274, 1996.
BADDELEY, Adrian , RUBAK, Ege, TURNER, Rolf Turner. Spatial Point Patterns: Methodology and Applications with R Chapman and Hall/CRC 810 Pages. 1st Edition 2015.
BIVAND, Roger. et al. Applied Spatial Data Analysis with R, Springler 2008