5 Visualizando dados com ggplot2

O ggplot2 é um pacote para visualização de dados criado por Hadley Wickham e parte do tidyverse. O ggplot2 implementa um sistema de gráficos baseados em uma gramática própria que permite a criação de gráficos avançados. Essa gramática se encarrega de diversos detalhes que precisariam ser especificados usando o módulo base do R e organiza um gráfico como a soma de camadas.
A grámatica do ggplot2 pode parecer mais complexa à primeira vista. Porém, após nos familiarizarmos com ela, conseguimos, em poucas linhas, criar gráficos sofisticados, compostos por múltiplas camadas e com excelente qualidade visual. Com ggplot2 é possível combinar elementos e facilmente criar novos gráficos de muitas maneiras customizando sua aparência. Atualmente podemos encontrar mais de 150 pacotes do R que estendem ou implementam funções gráficas do ggplot2. Podemos ver alguns destes pacotes AQUI.
Neste curso faremos uma breve introdução sobre o ggplot2 e seus tipos de gráficos mais comuns. Não cobriremos todas as possibilidades de visualização de dados com o ggplot2, que são muito extensas. Para ssaber mais sobre o ggplot2 e encontrar diversos materiais de referência, clique AQUI.
5.1 Camadas
O ggplot2 segue a lógica da Grammar of Graphics, em que um gráfico é construído pela soma de camadas. Cada camada adiciona uma parte à visualização final — e podemos empilhar quantas camadas quisermos usando o operador +.
Veja o exemplo abaixo utilizando dados de personagens de Star Wars presente no pacote dados:
library(dados)
dados_starwars
# A tibble: 87 × 14
nome altura massa cor_do_cabelo cor_da_pele cor_dos_olhos ano_nascimento sexo_biologico genero planeta_natal especie
<chr> <int> <dbl> <chr> <chr> <chr> <dbl> <chr> <chr> <chr> <chr>
1 Luke Skyw… 172 77 Loiro Branca cla… Azul 19 Macho Mascu… Tatooine Humano
2 C-3PO 167 75 NA Ouro Amarelo 112 Nenhum Mascu… Tatooine Droide
3 R2-D2 96 32 NA Branca, Az… Vermelho 33 Nenhum Mascu… Naboo Droide
4 Darth Vad… 202 136 Nenhum Branca Amarelo 41.9 Macho Mascu… Tatooine Humano
5 Leia Orga… 150 49 Castanho Clara Castanho 19 Fêmea Femin… Alderaan Humano
6 Owen Lars 178 120 Castanho, Ci… Clara Azul 52 Macho Mascu… Tatooine Humano
7 Beru Whit… 165 75 Castanho Clara Azul 47 Fêmea Femin… Tatooine Humano
8 R5-D4 97 32 NA Branca, Ve… Vermelho NA Nenhum Mascu… Tatooine Droide
9 Biggs Dar… 183 84 Preto Clara Castanho 24 Macho Mascu… Tatooine Humano
10 Obi-Wan K… 182 77 Ruivo, Branco Branca cla… Azul acinzen… 57 Macho Mascu… Stewjon Humano
# ℹ 77 more rows
# ℹ 3 more variables: filmes <list>, veiculos <list>, naves_espaciais <list>
# ℹ Use `print(n = ...)` to see more rowsggplot(data = dados_starwars, aes(x = altura, y = massa)) +
geom_point()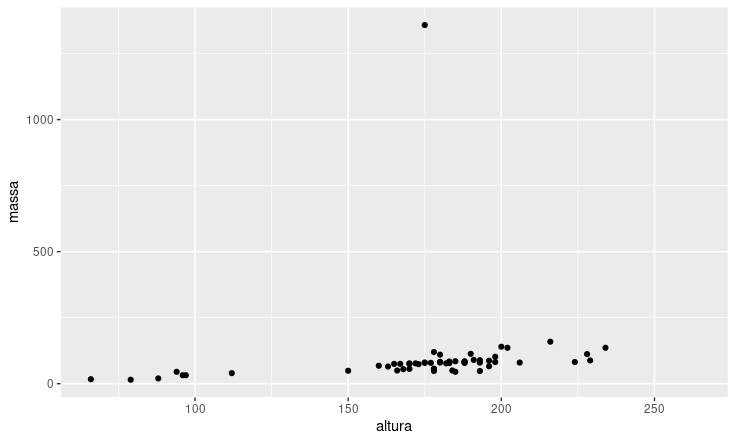
Esse código:
- Especifica o uso do dataset
dados_starwarsemggplot()
- Mapeia a estética em
aes()especificando quais as variáveisalturapara o eixo x emassapara o eixo y
- Soma uma camada com o operador
+
- Usa geom_point() para adicionar pontos ao gráfico.
Esses componentes são os mínimos obrigatórios para gerar qualquer gráfico com o ggplot2: dados, mapeamentos estéticos, operador + e geometria (geom_*()).
Uma vez definido o básico, podemos adicionar camadas para refinar e customizar o gráfico com camadas opcionais:
ggplot(dados_starwars, aes(x = altura, y = massa, color = sexo_biologico)) +
geom_point() +
labs(
title = "Relação entre altura e massa entre personagens de Star Wars",
x = "Altura (cm)",
y = "Massa (kg)"
) +
annotate(
geom = "text",
label = "Jabba the Hutt",
x = 195,
y = 1350
) +
theme_minimal()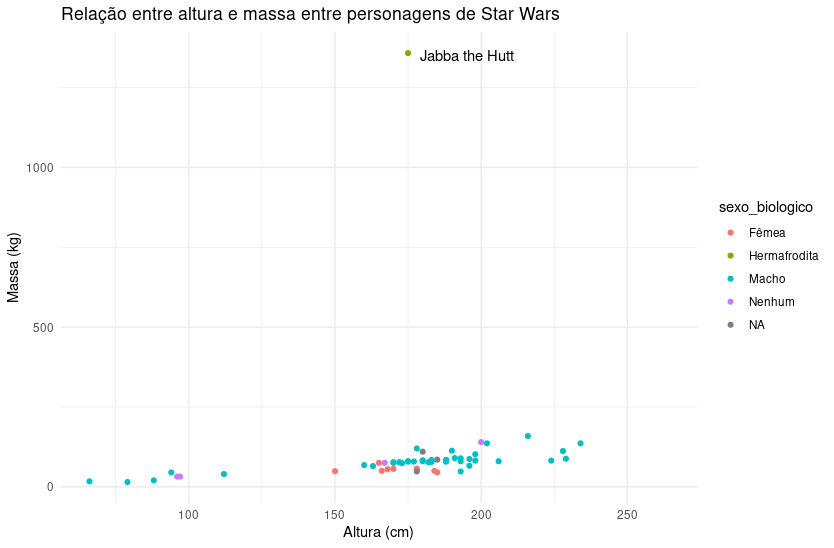
Agora na estética especificamos que as observações sejam coloridas de acordo com o sexo biológico. Além disso, adicionamos título e nome dos eixos x e y dentro do labs() e com anottate() adicionamos uma anotação com o nome do personagem no ponto outlier. Além disso, utilizamos o tema theme_minimal() para estilizar o gráfico.
Este é apenas um exemplo! Existem muitas outras possibilidades!
Veja abaixo um resumo dos principais componentes utilizados para criar gráficos no ggplot2:
| Componente (Camada) | Descrição | Funções principais |
|---|---|---|
| Dados | Conjunto de dados que será visualizado. | ggplot(data = ...) |
| Mapeamentos estéticos | Define como variáveis são mapeadas para aspectos visuais (x, y, cor, forma, tamanho). | aes(x = ..., y = ..., color = ..., size = ...) |
| Geometria | Tipo de representação visual a ser usada. | geom_point(), geom_line(), geom_bar(), geom_histogram(), geom_boxplot(), geom_sf() |
| Transformações estatísticas | Calcula estatísticas antes de desenhar (por exemplo, contagens ou regressões). | stat_summary(), stat_smooth(), stat_bin() (muitas geoms já têm stat implícito) |
| Escalas | Controla eixos, cores, tamanhos, formas. Pode mudar intervalos, rótulos e paletas. | scale_x_date(), scale_y_continuos(), scale_color_brewer(), scale_fill_manual() |
| Facetas | Divide o gráfico em múltiplos painéis para comparação. | facet_wrap(~variavel), facet_grid(linhas ~ colunas) |
| Rótulos | Adiciona ou personaliza títulos, subtítulos, legendas. | labs(), ggtitle(), xlab(), ylab() |
| Temas | Ajusta a aparência do gráfico (fonte, fundo, grade, posição da legenda). | theme_minimal(), theme_bw(), theme() |
| Coordenadas | Controla sistema de coordenadas e projeções. | coord_cartesian(), coord_flip(), coord_polar() |
| Anotações | Adiciona textos, formas ou linhas extras ao gráfico. | annotate("text", ...), annotate("segment", ...), geom_hline(), geom_vline() |
5.2 A camada estética aes()
Vamos conhecer um pouco mais as opções disponíveis na camada estética (aes()). Nela, você pode definir diferentes parâmetros:
| Parâmetro | O que controla |
|---|---|
x |
Variável para o eixo x |
y |
Variável para o eixo y |
color ou colour |
Cor da linha, ponto ou contorno, mapeada a uma variável |
fill |
Cor de preenchimento de áreas ou barras, mapeada a uma variável |
size |
Tamanho de pontos proporcional a uma variável |
linewidth |
Espessura de linhas proporcional a uma variável |
shape |
Forma dos pontos, diferenciada por categorias de uma variável |
linetype |
Tipo de linha (contínua, tracejada, pontilhada) de acordo com uma variável |
alpha |
Transparência (0 = invisível, 1 = opaco) mapeada a uma variável numérica |
group |
Agrupa observações para conectar linhas ou desenhar polígonos corretamente |
label |
Texto exibido em geoms que desenham rótulos (geom_text(), geom_label()) |
Alguns desses parâmetros podem ser definidos fora de aes(). Qual a diferença?
Perceba que no exemplo anterior especificamos color = sexo_biologico dentro de aes() para que os pontos fossem coloridos de acordo com essa variável. Dentro de aes() especificamos a estética a partir de variáveis. Fora de aes(), definimos valores fixos.
Vamos voltar ao exemplo de massa vs altura dos personagens de Star Wars e modificar o formato (shape) das observações. Os formatos podem assumir os seguintes valores:
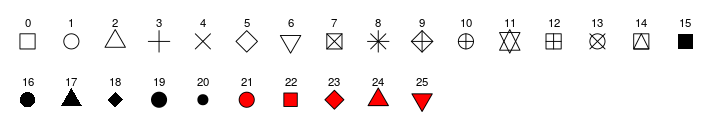
Nota: Para os formatos sólidos de 21 a 25 pode-se definir tanto a cor da linha com color quanto o preenchimento com fill.
Primeiro, vamos pedir que o formato varie de acordo com o sexo biológico:
ggplot(dados_starwars, aes(x = altura,
y = massa,
color = sexo_biologico,
shape = sexo_biologico)) +
geom_point() 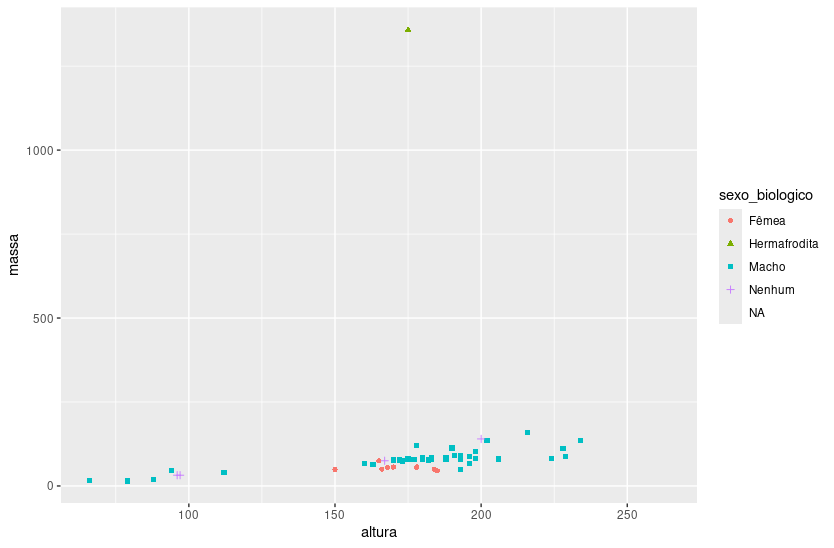
Perceba que automaticamente o ggplot2 cria a legenda combinando os dois parâmetros especificados, cor e formato.
Agora vamos supor que eu queira todos com cor azul. Veja o que acontece se especifico dentro de aes():
ggplot(dados_starwars, aes(x = altura,
y = massa,
color = 'blue',
shape = sexo_biologico)) +
geom_point() 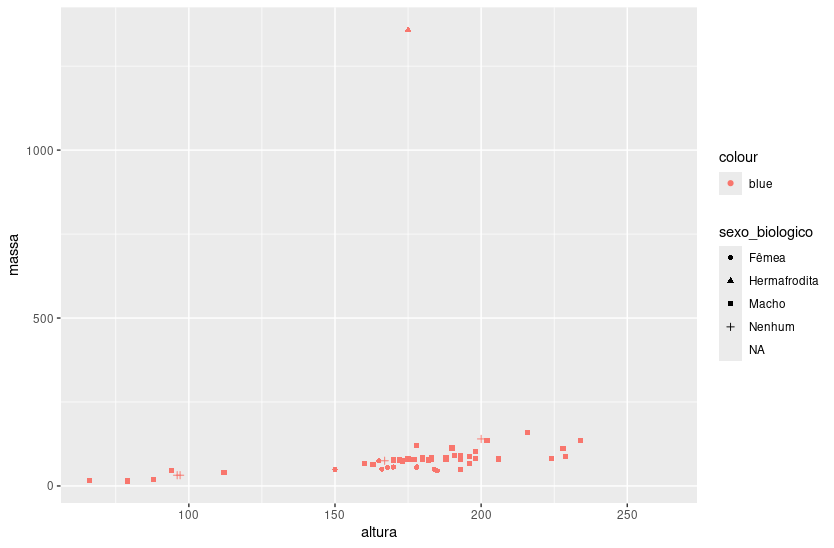
O que aconteceu?
Como corrigimos?
ggplot(dados_starwars,
aes(x = altura,
y = massa,
shape = sexo_biologico),
color = 'blue') +
geom_point() 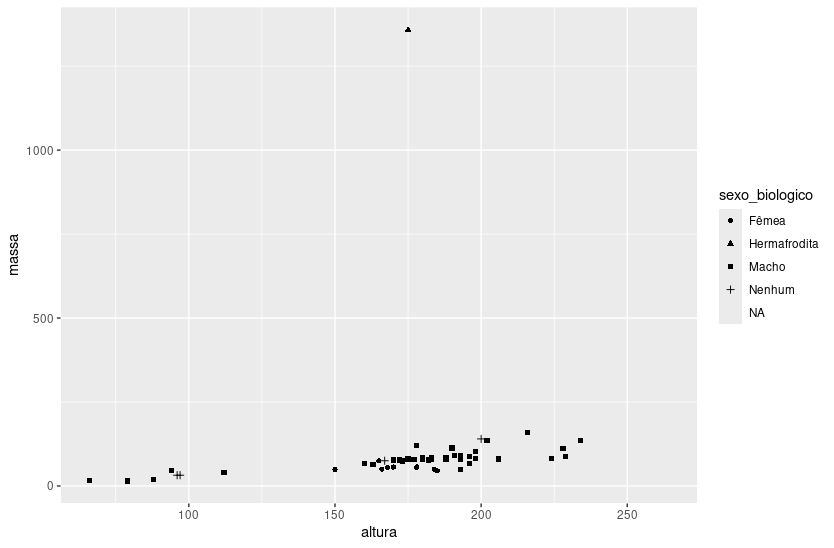
Funcionou?
No ggplot2, quando você define um argumento de estética fora do aes(), ele precisa estar na mesma camada onde você quer aplicar o estilo.
ggplot(dados_starwars, aes(x = altura,
y = massa,
shape = sexo_biologico)) +
geom_point(color = 'blue') 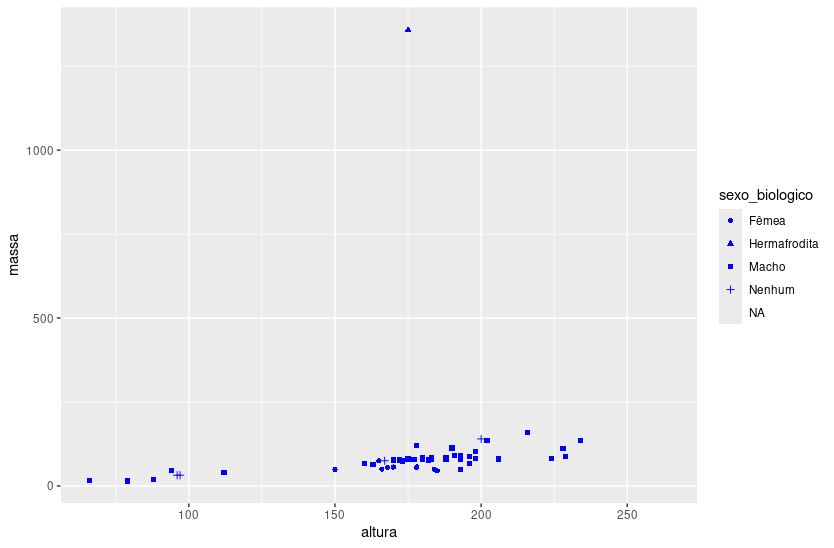
Agora vamos voltar aos dados de AIDS no Brasil. Os dados arrumados e agrupados por UF de residência e ano podem ser baixados aqui:
url <- "https://raw.githubusercontent.com/laispfreitas/curso_CD1/refs/heads/main/aids_uf.csv"
aids.uf <- read_csv(url)
aids.uf
# A tibble: 1,188 × 7
ano cod_UF_res casos abbrev_state name_state code_region name_region
<dbl> <dbl> <dbl> <chr> <chr> <dbl> <chr>
1 1980 11 0 RO Rondônia 1 Norte
2 1980 12 0 AC Acre 1 Norte
3 1980 13 0 AM Amazonas 1 Norte
4 1980 14 0 RR Roraima 1 Norte
5 1980 15 0 PA Pará 1 Norte
6 1980 16 0 AP Amapá 1 Norte
7 1980 17 0 TO Tocantins 1 Norte
8 1980 21 0 MA Maranhão 2 Nordeste
9 1980 22 0 PI Piauí 2 Nordeste
10 1980 23 0 CE Ceará 2 Nordeste
# ℹ 1,178 more rows
# ℹ Use `print(n = ...)` to see more rowsVamos agora fazer um gráfico com as séries temporais das UFs do Nordeste, variando tanto a cor quanto o tipo de linha.
Os tipos de linha podem ser os seguintes:

Vamos selecionar as UFs desejadas:
aids.NE <- aids.uf |>
filter(name_region == "Nordeste")
aids.NE
# A tibble: 396 × 7
ano cod_UF_res casos abbrev_state name_state code_region name_region
<dbl> <dbl> <dbl> <chr> <chr> <dbl> <chr>
1 1980 21 0 MA Maranhão 2 Nordeste
2 1980 22 0 PI Piauí 2 Nordeste
3 1980 23 0 CE Ceará 2 Nordeste
4 1980 24 0 RN Rio Grande Do Norte 2 Nordeste
5 1980 25 0 PB Paraíba 2 Nordeste
6 1980 26 0 PE Pernambuco 2 Nordeste
7 1980 27 0 AL Alagoas 2 Nordeste
8 1980 28 0 SE Sergipe 2 Nordeste
9 1980 29 0 BA Bahia 2 Nordeste
10 1982 21 0 MA Maranhão 2 Nordeste
# ℹ 386 more rows
# ℹ Use `print(n = ...)` to see more rowsgg_aidsNE <- ggplot(aids.NE, aes(x = ano,
y = casos,
color = abbrev_state,
linetype = abbrev_state)) +
geom_line(linewidth = 1)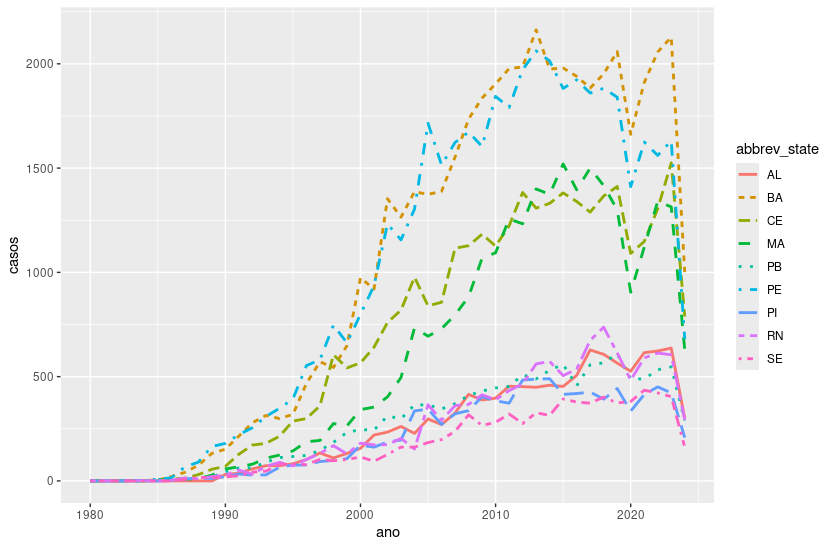
Perceba que adicionamos linewidth = 1 em geom_line() para especificar a espessura da linha.
5.3 Temas
Os temas do ggplot2 são o que controlam a aparência não relacionada aos dados do gráfico, como:
- Cor de fundo do gráfico e do painel de plotagem
- Linhas de grade (major/minor gridlines)
- Títulos e textos (fonte, tamanho, posição)
- Aparência de legendas (posição, caixa, título)
- Margens e espaçamentos
Os temas podem ser adicionados ao ggplot como uma nova camada, com o operador +, como já vimos em exemplos passados. Se um tema não for adicionado, o ggplot2 usará o seu tema padrão, theme_gray().
Existem outros temas no pacote ggplot2 que podem ser utilizados: theme_bw(), theme_minimal(), theme_light(), theme_dark(), theme_classic() e theme_void().
Vamos refazer o gráfico anterior usando o theme_light():
ggplot(aids.NE, aes(x = ano,
y = casos,
color = abbrev_state,
linetype = abbrev_state)) +
geom_line(linewidth = 1) +
theme_light()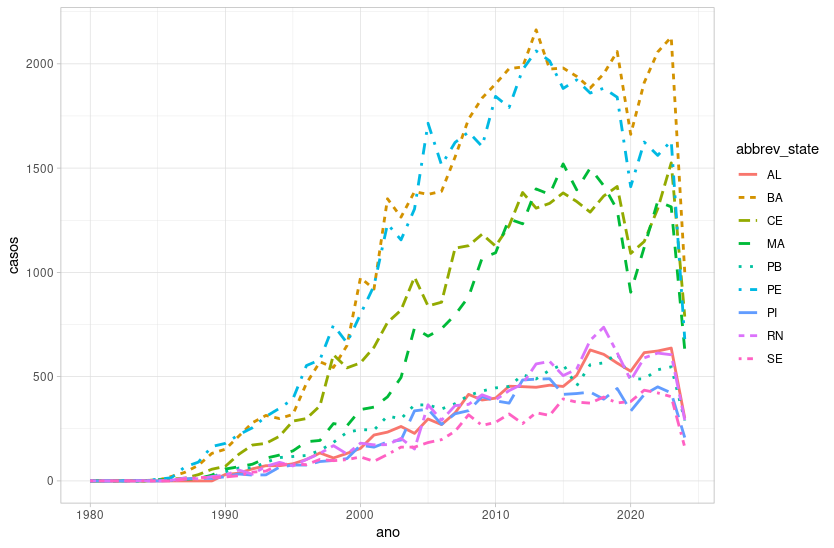
Além de usar temas prontos, você pode personalizar elementos específicos com theme():
ggplot(aids.NE, aes(x = ano,
y = casos,
color = abbrev_state,
linetype = abbrev_state)) +
geom_line(linewidth = 1) +
labs(title = "Casos de AIDS registrados no Nordeste por UF",
x = "Ano",
y = "Número de casos",
color = "UF",
linetype = "UF") +
theme(text = element_text(size = 16),
plot.title = element_text(hjust = .5, face = "bold"),
legend.position = "bottom",
axis.line = element_line(color = 'black'),
panel.grid.major = element_line(color = 'grey90', linewidth = .3),
panel.background = element_rect(fill = 'white'))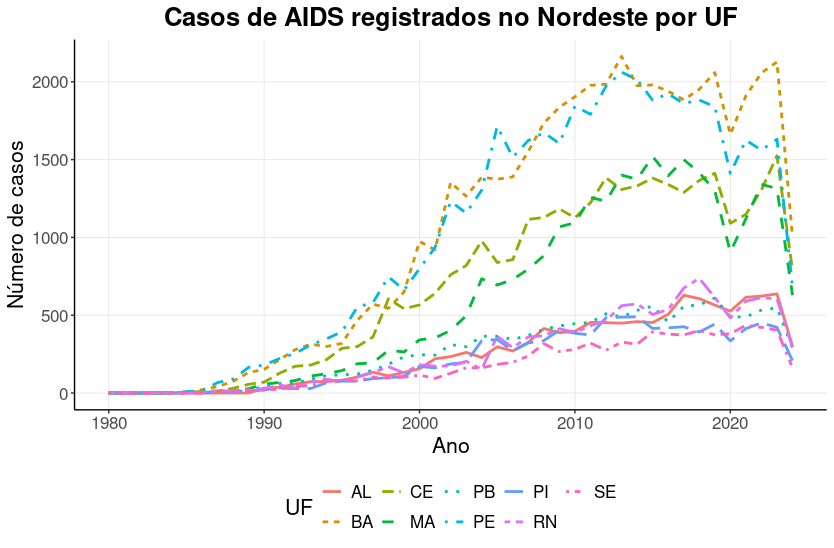
Alguns pacotes oferecem opções de temas adicionais para o ggplot2. Um dos mais famosos é o ggthemes, que tem temas baseados em estilos de jornais ou publicações famosas.
library(ggthemes)
ggplot(aids.NE, aes(x = ano,
y = casos,
color = abbrev_state,
linetype = abbrev_state)) +
geom_line(linewidth = 1) +
labs(title = "Casos de AIDS registrados no Nordeste por UF",
x = "Ano",
y = "Número de casos",
color = "UF",
linetype = "UF") +
theme_clean() 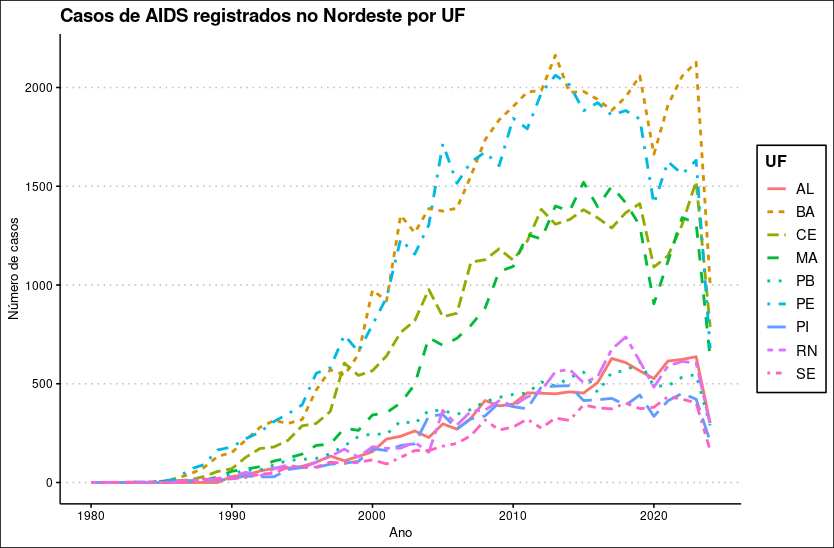
5.4 Facetas
Podemos separar os gráficos para que cada UF fique em uma faceta acrescentando uma camada com facet_wrap():
ggplot(aids.NE, aes(x = ano,
y = casos,
color = abbrev_state,
linetype = abbrev_state)) +
geom_line(linewidth = 1) +
facet_wrap(~abbrev_state)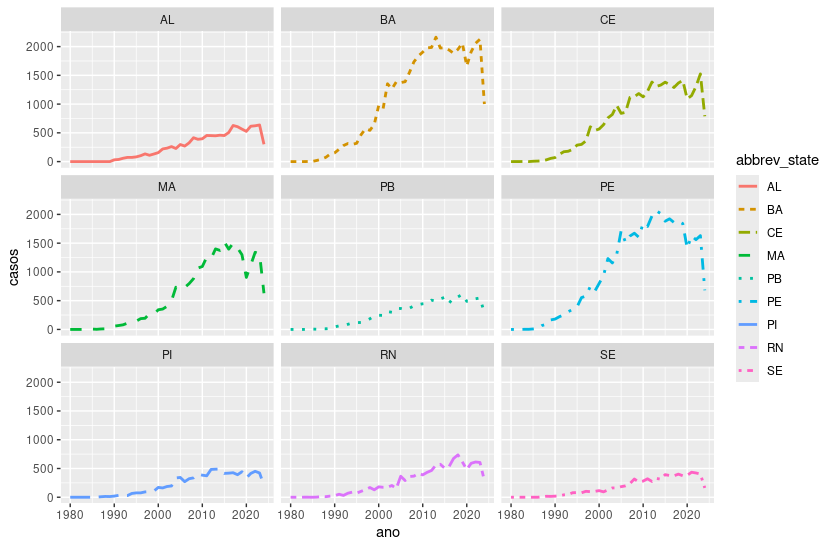
No exemplo acima todos as facetas ficaram na mesma escala no eixo x e y. Em alguns casos pode ser interessante deixar o eixo y variar. Isto pode ser especificado acrescentando scale = "free_y" em facet_wrap().
ggplot(aids.NE, aes(x = ano,
y = casos,
color = abbrev_state,
linetype = abbrev_state)) +
geom_line(linewidth = 1) +
facet_wrap(~abbrev_state, scale = "free_y")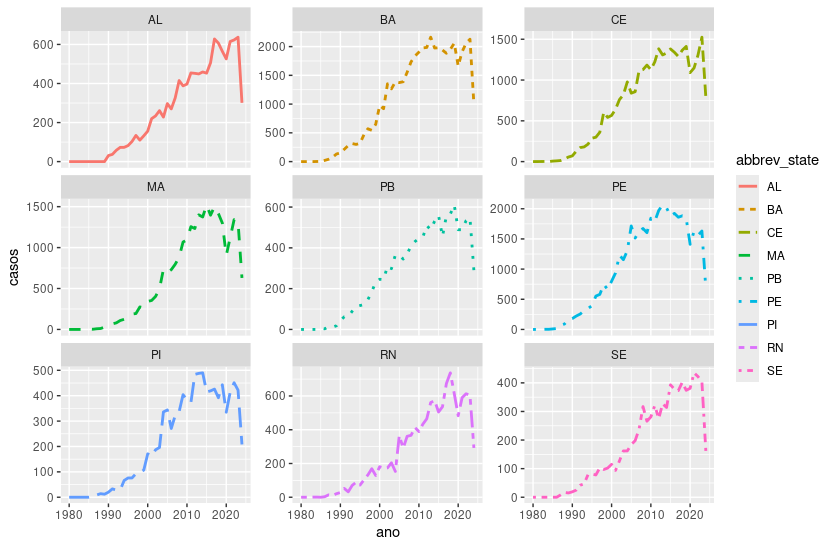
5.4.1 facet_geo

Existe um pacote chamado geofacet que permite adicionar uma camada com facet_geo() ao ggplot. As facetas resultantes preservação em parte a orientação geográfica. Para saber mais sobre a geofacet, clique AQUI.
Como temos os dados de AIDS por UF, podemos usar esse pacote para ter as facetas preservando em parte a localização das UFs no Brasil.
Agora vamos fazer o gráfico:
library(geofacet)
ggplot(aids.uf2, aes(x = ano,
y = casos,
color = abbrev_state)) +
geom_line(linewidth = 1) +
facet_geo(~abbrev_state, grid = "br_states_grid1")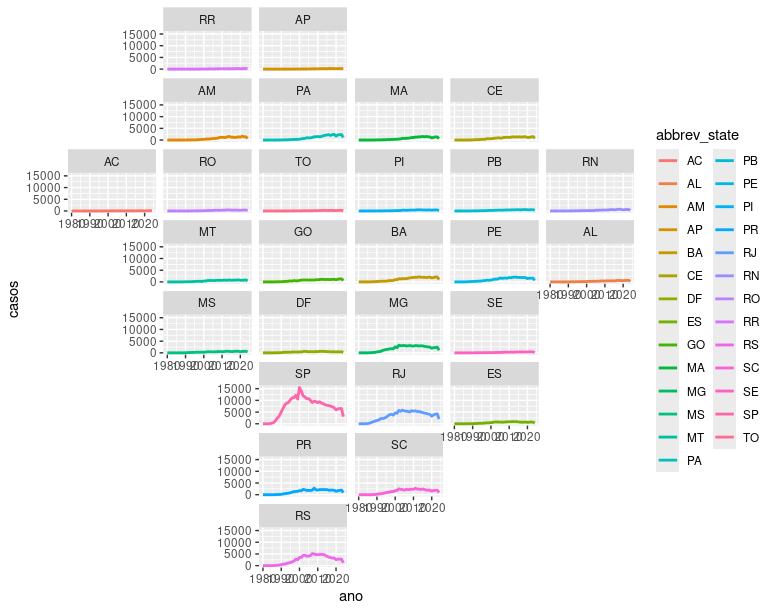
Legal, mas vamos melhorar esse gráfico? O que podemos fazer?
5.5 Histogramas e densidade
Agora que já vimos a ideia de camadas e conhecemos as opções de mapeamento estético em aes(), vamos fazer um histograma:
ggplot(iris, aes(x = Sepal.Width)) +
geom_histogram()
`stat_bin()` using `bins = 30`. Pick better value with `binwidth`.Repare que retornou uma mensagem de aviso. Podemos especificar a quantidade de classes usando os parâmetros binwidth ou bins. Você deve sempre buscar um valor mais adequado que o padrão (que é de 30 classes).
p <- ggplot(iris, aes(x = Sepal.Width))
p + geom_histogram(binwidth = 0.1)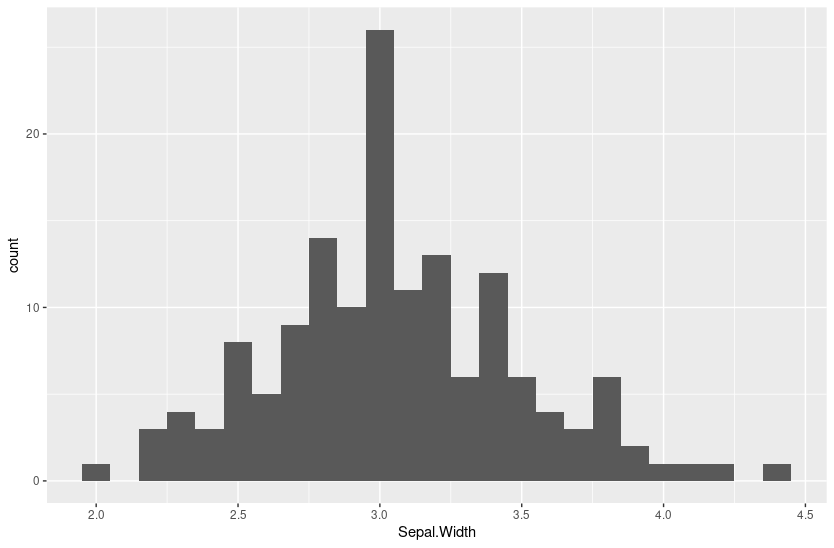
Alterando o número de classes:
p + geom_histogram(bins = 20)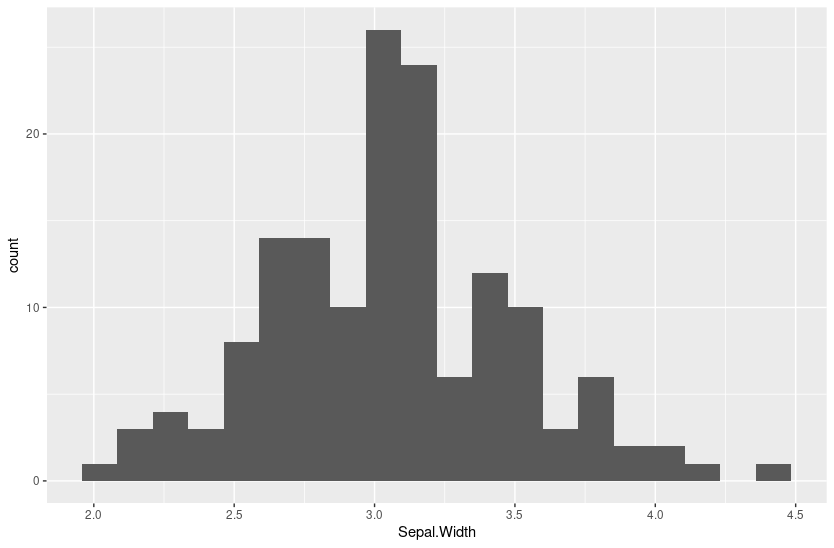
Existe uma fórmula para converter aproximadamente bins em binwidth. Considerando bins = 20:
diff(range(iris$Sepal.Width) / 20)
[1] 0.12Suponha agora que queremos um histograma para cada especie.
ggplot(iris, aes(Sepal.Width, fill = Species)) +
geom_histogram(binwidth = 0.1)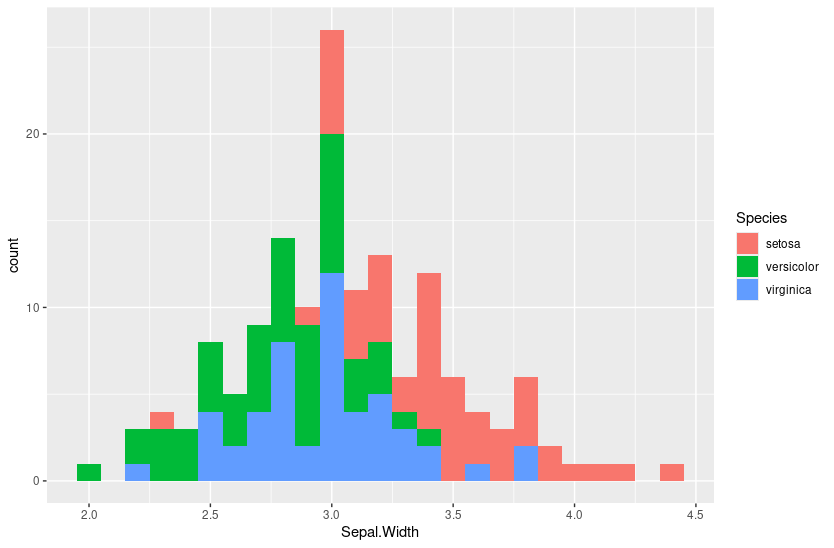
Podemos também usar o parâmetro position em geom_histogram() para especificar barras lado a lado ou empilhadas. Teste as opções dodge e fill. A padrão é stack.
ggplot(iris, aes(Sepal.Width, fill = Species)) +
geom_histogram(binwidth = 0.1, position = "dodge")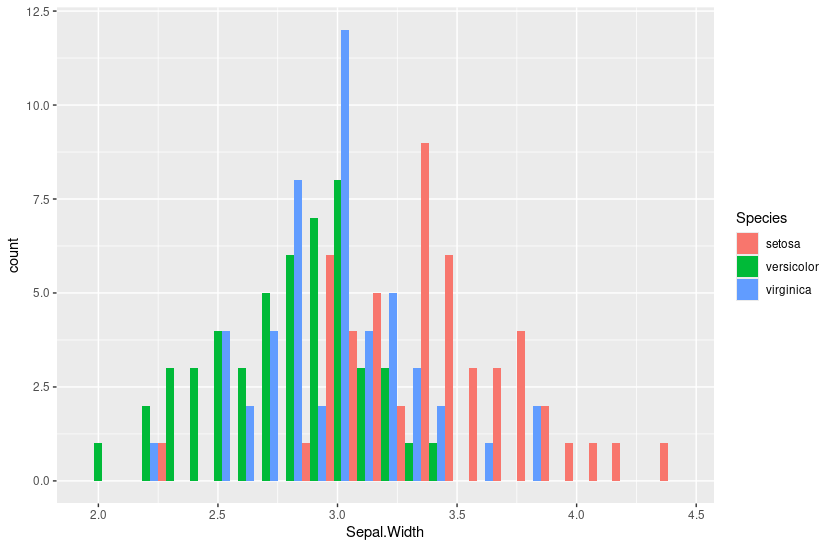
Teste também as opções de coordenadas cord_flip():
ggplot(iris, aes(Sepal.Width, fill = Species)) +
geom_histogram(binwidth = 0.1) +
coord_flip() 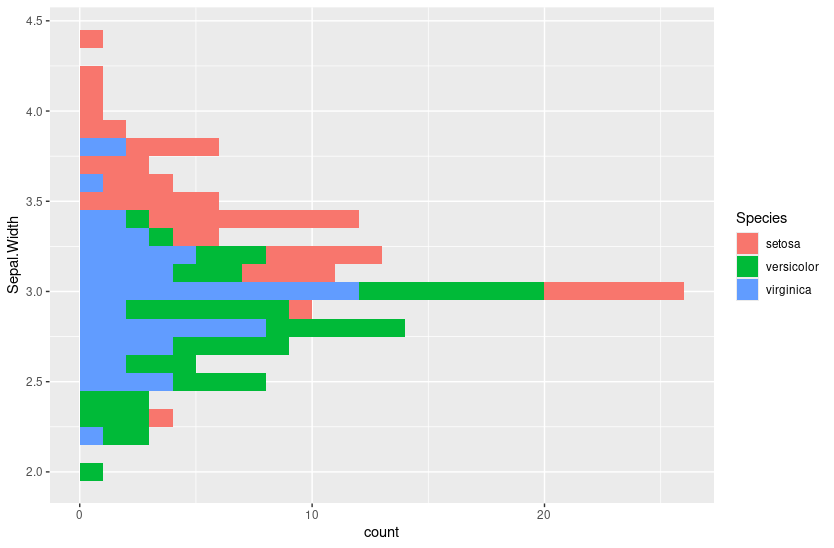
E coord_polar():
ggplot(iris, aes(Sepal.Width, fill = Species)) +
geom_histogram(binwidth = 0.1) +
coord_polar() 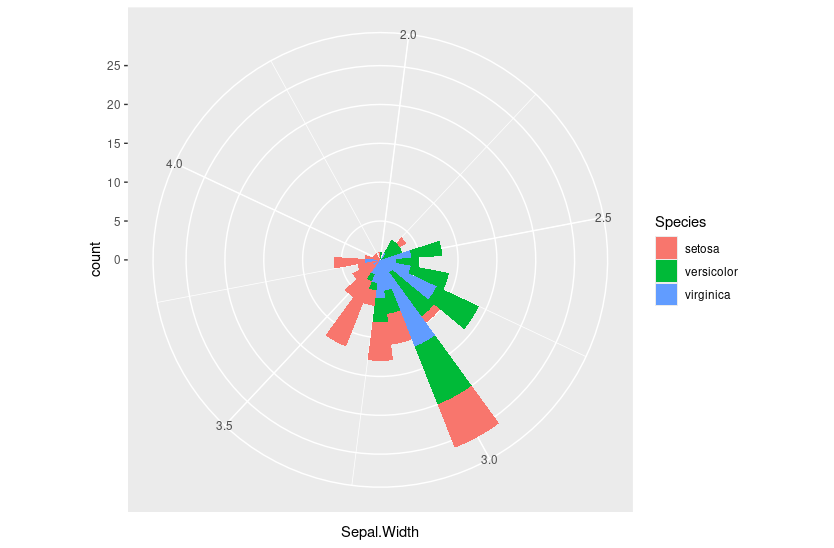
A função geom_density() é semelhante. Vamos testar com o parâmetro alpha:
ggplot(iris, aes(Sepal.Width, fill = Species)) +
geom_density(binwidth = 0.1, alpha = 0.6) 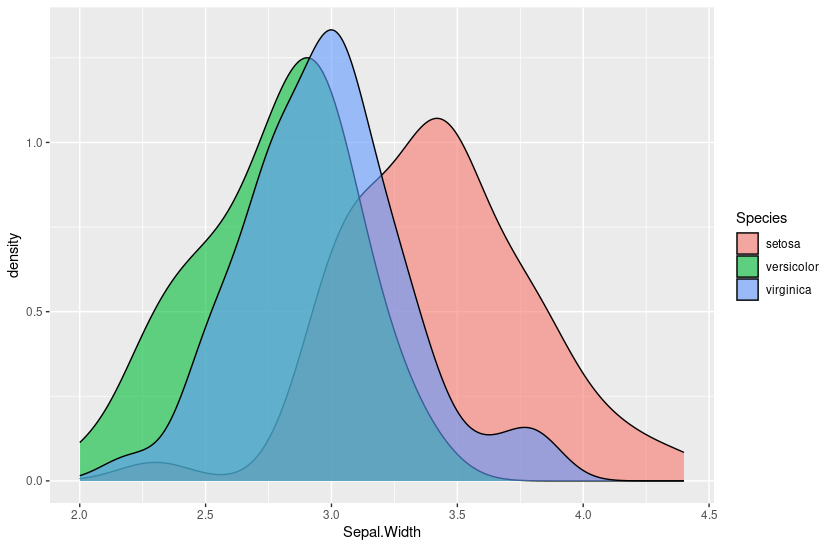
5.6 Gráfico de barras (barplot)
Um gráfico de barras pode ser facilmente obtido com geom_bar(). Por padrão, o parâmetro stat = "count", ou seja, o eixo y mostrará a quantidade de ocorrências de cada tipo de categoria em x. Veja abaixo:
ggplot(msleep, aes(vore)) +
geom_bar() 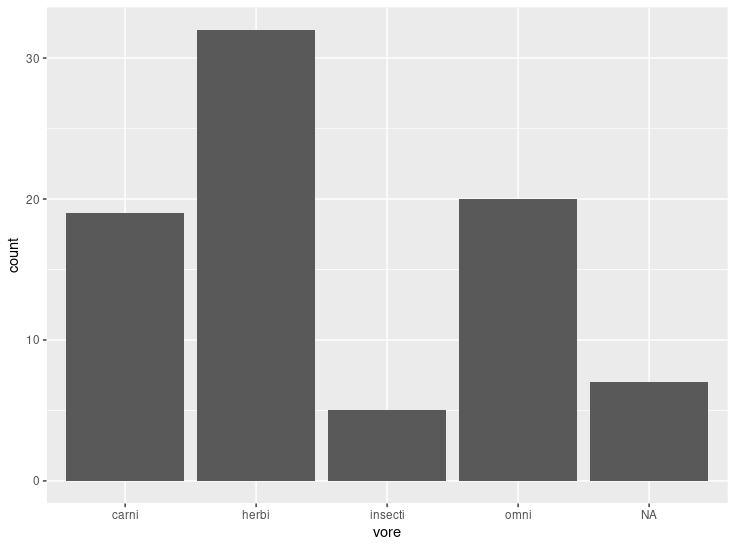
Agora vamos pra dados reais! Vamos utilizar dados que foram obtidos do Painel de Vigilância Genômica de Arboviroses do Ministério da Saúde com o número de amostras positivas para cada genótipo de vírus dengue em 2024 no Brasil.
url <- "https://raw.githubusercontent.com/laispfreitas/curso_CD1/refs/heads/main/tabela_dengue_genotipos.csv"
dengue <- read_csv(url) |>
janitor::clean_names()
# A tibble: 4 × 10
sorotipo em_branco denv_1_genotipo_v denv_2_genotipo_ii denv_2_genotipo_ii_cosmopolita denv_2_genotipo_iii_as…¹
<chr> <dbl> <dbl> <dbl> <dbl> <dbl>
1 DENV1 3 1738 NA NA NA
2 DENV2 21 NA NA 1405 58
3 DENV3 NA NA 1 NA NA
4 DENV4 NA NA NA NA NA
# ℹ abbreviated name: ¹denv_2_genotipo_iii_asiatico_americano
# ℹ 4 more variables: denv_2_genotipo_v_asiatico_i <dbl>, denv_3_genotipo_iii <dbl>, denv_4_genotipo_ii <dbl>,
# total <dbl>Observação: No código acima encadeamos a leitura do dado com a função clean_names() do pacote janitor para “limpar” os nomes das variáveis tornando-os únicos, todos em minículos e sem espaços em branco.
Vamos tentar fazer um barplot desses dados!
ggplot(dengue, aes(sorotipo)) +
geom_bar()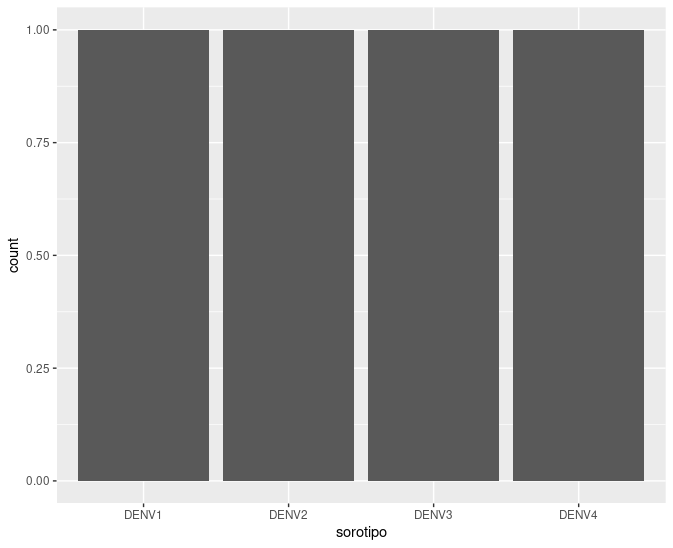
Funcionou?
Vamos pra segunda tentativa, então:
ggplot(dengue, aes(total)) +
geom_bar()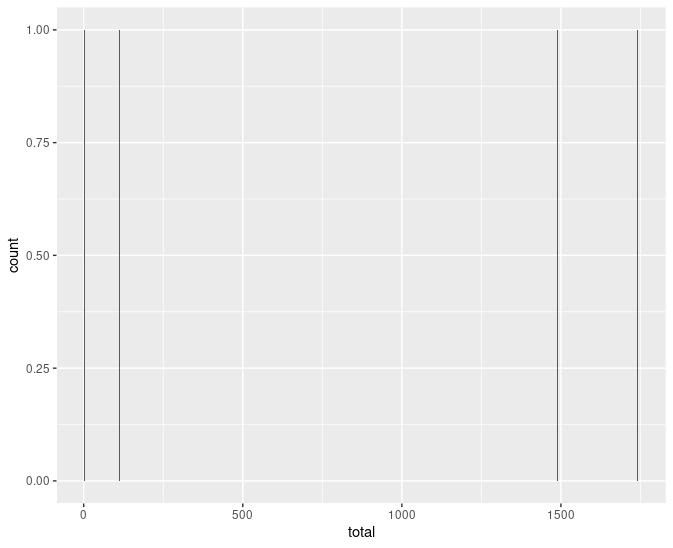
Ainda não funcionou!
Parece que vamos precisar das variável sorotipo e total.
ggplot(dengue, aes(sorotipo, total)) +
geom_bar()
Error in `geom_bar()`:
! Problem while computing stat.
ℹ Error occurred in the 1st layer.
Caused by error in `setup_params()`:
! `stat_count()` must only have an x or y aesthetic.
Run `rlang::last_trace()` to see where the error occurred.Continuamos sem sucesso, mas a mensagem de erro nos diz que função geom_bar() está contando o número de vezes que cada sorotipo aparece nos dados, então precisamos trocar o padrão stat = "count" para stat = "identity".
ggplot(dengue, aes(sorotipo, total, fill = sorotipo)) +
geom_bar(stat = "identity")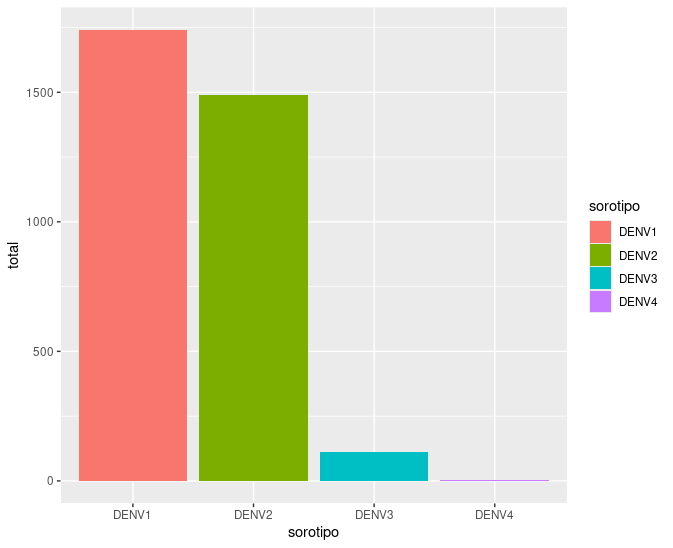
Ou seja:
Você deve usar stat = "count" para contar automaticamente o número de observações em cada categoria de x; e você deve usar stat = "identity" para usar diretamente os valores de uma variável em y.
Agora vamos representar no gráfico também as informações de genótipos, além de sorotipos. Perceba que os dados estão em formato largo (wide):
dengue
# A tibble: 4 × 10
sorotipo em_branco denv_1_genotipo_v denv_2_genotipo_ii denv_2_genotipo_ii_cosmopolita denv_2_genotipo_iii_as…¹
<chr> <dbl> <dbl> <dbl> <dbl> <dbl>
1 DENV1 3 1738 NA NA NA
2 DENV2 21 NA NA 1405 58
3 DENV3 NA NA 1 NA NA
4 DENV4 NA NA NA NA NA
# ℹ abbreviated name: ¹denv_2_genotipo_iii_asiatico_americano
# ℹ 4 more variables: denv_2_genotipo_v_asiatico_i <dbl>, denv_3_genotipo_iii <dbl>, denv_4_genotipo_ii <dbl>,
# total <dbl>O ggplot2 funciona melhor com dados no formato longo (long), porque ele mapeia variáveis para estéticas (aes()) — se os genótipos são colunas, você não consegue dizer fill = genotipo (não existe uma coluna com esse nome!).
Então vamos precisar usar uma função do tidyr que vimos no capítulo 4! Qual é ela?
dengue.longo <- dengue |>
select(-total) |>
pivot_longer(
cols = -sorotipo,
values_to = 'n',
names_to = 'genotipo'
)
dengue.longo
# A tibble: 32 × 3
sorotipo genotipo n
<chr> <chr> <dbl>
1 DENV1 em_branco 3
2 DENV1 denv_1_genotipo_v 1738
3 DENV1 denv_2_genotipo_ii NA
4 DENV1 denv_2_genotipo_ii_cosmopolita NA
5 DENV1 denv_2_genotipo_iii_asiatico_americano NA
6 DENV1 denv_2_genotipo_v_asiatico_i NA
7 DENV1 denv_3_genotipo_iii NA
8 DENV1 denv_4_genotipo_ii NA
9 DENV2 em_branco 21
10 DENV2 denv_1_genotipo_v NA
# ℹ 22 more rows
# ℹ Use `print(n = ...)` to see more rowsPronto! Agora podemos fazer nosso gráfico!
ggplot(dengue.longo, aes(x = sorotipo, y = n, fill = genotipo)) +
geom_bar(stat = "identity")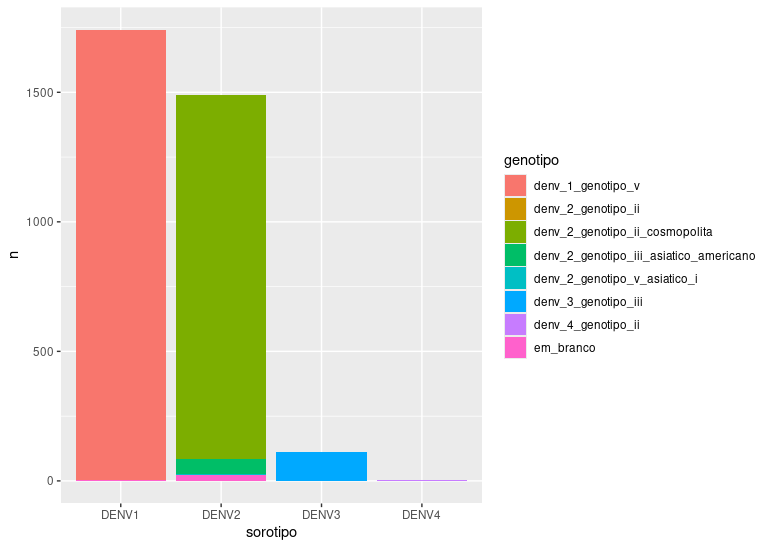
Por padrão, as barras aparecerão empilhadas, ou seja, position = "stack". Podemos usar outras opções.
position |
O que faz | Exemplo visual |
|---|---|---|
"stack" (padrão) |
Empilha os valores um em cima do outro (mostra soma total). | Barras empilhadas |
"dodge" |
Coloca lado a lado as barras de cada grupo. | Barras lado a lado |
"fill" |
Igual a stack, mas normaliza para 100% (proporções). | Barras empilhadas com mesma altura |
"identity" |
Usa as alturas exatamente como estão, sem empilhar. Pode sobrepor barras. | Cada barra fica independente (pode sobrepor) |
Vamos testar position = "fill":
ggplot(dengue.longo, aes(x = sorotipo, y = n, fill = genotipo)) +
geom_bar(stat = "identity", position = "fill")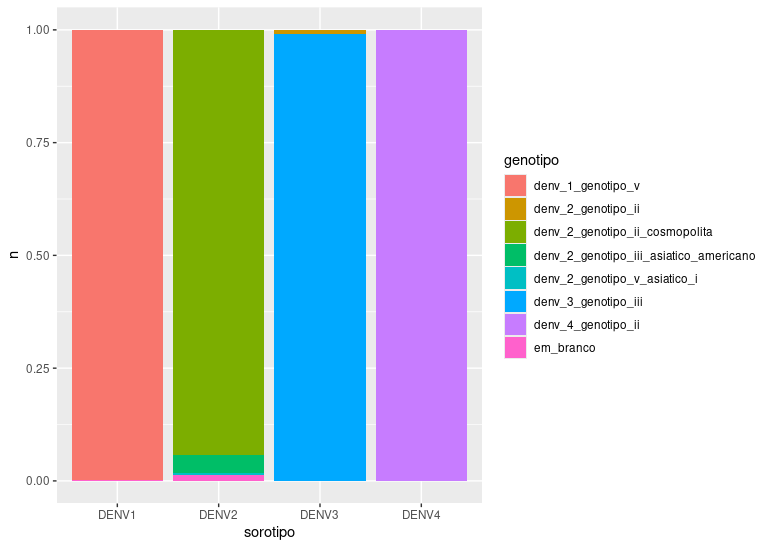
O que aconteceu?
Agora teste outras opções de position =, escolha uma e finalize o gráfico usando labs() para acrescentar um título, definir os rótulos de x, y, e o título da legenda, e adicione um tema.
5.7 Boxplot
Um boxplot é um dos gráficos mais comuns para explorar a distribuição de uma variável numérica. Pode ser obtido com o ggplot() mais a camada geom_boxplot().
Para explorar esse tipo de gráfico, vamos voltar aos dados de AIDS no Brasil:
aids.uf
# A tibble: 1,188 × 7
ano cod_UF_res casos abbrev_state name_state code_region name_region
<dbl> <dbl> <dbl> <chr> <chr> <dbl> <chr>
1 1980 11 0 RO Rondônia 1 Norte
2 1980 12 0 AC Acre 1 Norte
3 1980 13 0 AM Amazonas 1 Norte
4 1980 14 0 RR Roraima 1 Norte
5 1980 15 0 PA Pará 1 Norte
6 1980 16 0 AP Amapá 1 Norte
7 1980 17 0 TO Tocantins 1 Norte
8 1980 21 0 MA Maranhão 2 Nordeste
9 1980 22 0 PI Piauí 2 Nordeste
10 1980 23 0 CE Ceará 2 Nordeste
# ℹ 1,178 more rows
# ℹ Use `print(n = ...)` to see more rowsPrimeiro, vamos agrupar os dados por região e ano.
aids.regiao <- aids.uf |>
group_by(ano, name_region) |>
summarise(casos = sum(casos, na.rm = TRUE))
aids.regiao
# A tibble: 220 × 3
# Groups: ano [44]
ano name_region casos
<dbl> <chr> <dbl>
1 1980 Centro Oeste 0
2 1980 Nordeste 0
3 1980 Norte 0
4 1980 Sudeste 1
5 1980 Sul 0
6 1982 Centro Oeste 0
7 1982 Nordeste 1
8 1982 Norte 0
9 1982 Sudeste 14
10 1982 Sul 2
# ℹ 210 more rows
# ℹ Use `print(n = ...)` to see more rowsO dado já está em formato longo, então vamos partir para o ggplot!
ggplot(aids.regiao, aes(name_region, casos)) +
geom_boxplot() +
theme_linedraw()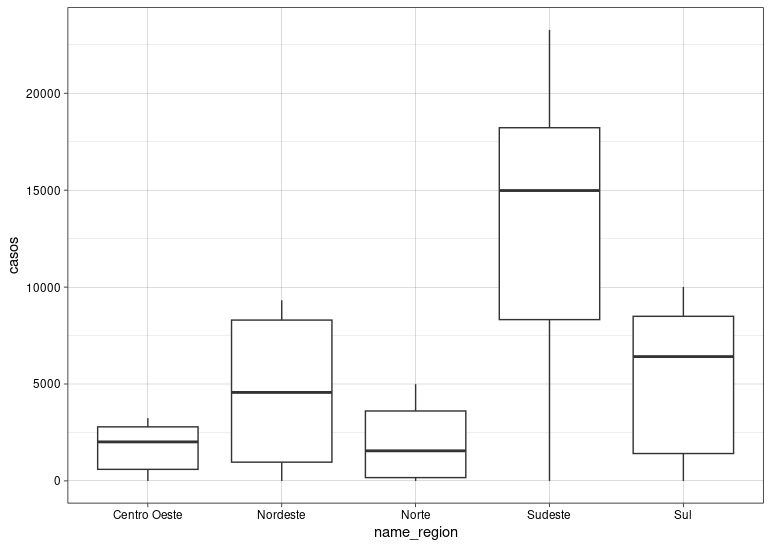
Poderia rodar as coordenadas e colorir por região:
ggplot(aids.regiao, aes(name_region, casos, fill = name_region)) +
geom_boxplot() +
coord_flip() +
labs(title = "AIDS no Brasil por região, 1980-2024*",
caption = "*Dados de 2024 são preliminares",
y = "Número de casos",
x = "Região",
fill = "Região") +
theme_linedraw()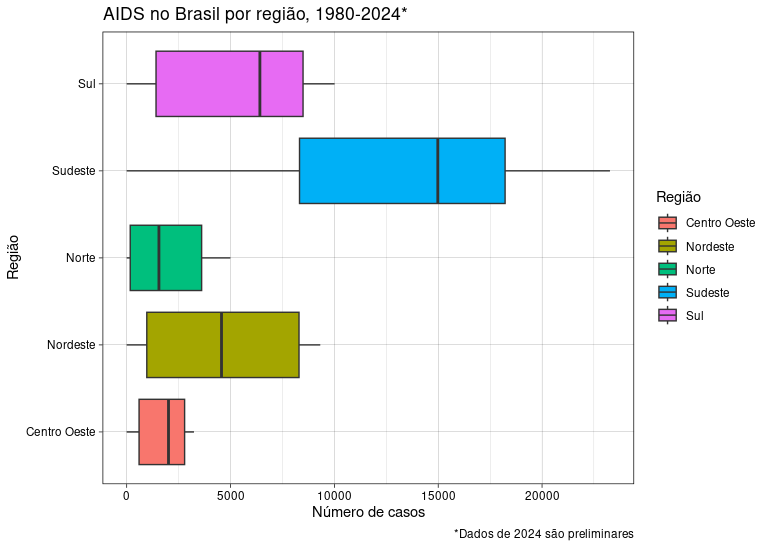
Podemos acrescentar os pontos dos valores individuais de forma aleatória acrescentando uma camada de geom_jitter():
ggplot(aids.regiao, aes(name_region, casos, fill = name_region)) +
geom_boxplot() +
geom_jitter(width = 0.2, alpha = 0.3) +
coord_flip() +
labs(title = "AIDS no Brasil por região, 1980-2024*",
caption = "*Dados de 2024 são preliminares",
y = "Número de casos",
x = "Região",
fill = "Região") +
theme_linedraw()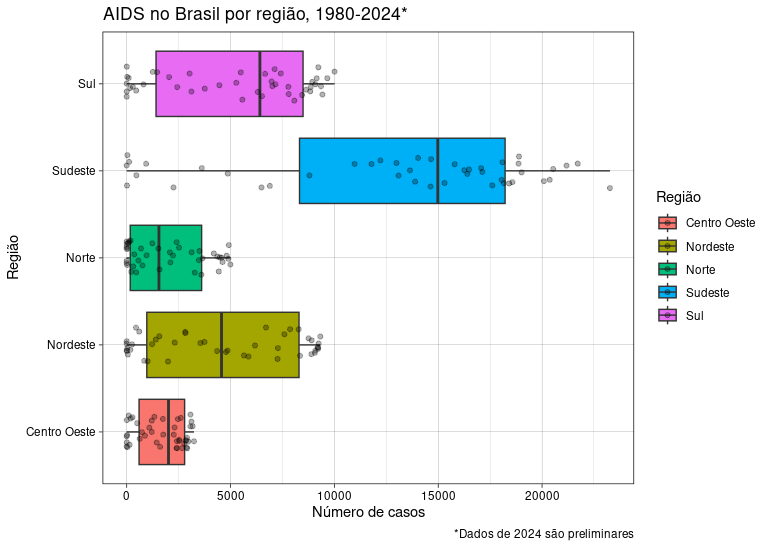
E se quisermos ver a distribuição de casos a cada década?
aids.dec <- aids.uf |>
mutate(decada = case_when(
ano < 1990 ~ '1980s',
ano < 2000 ~ '1990s',
ano < 2010 ~ '2000s',
ano < 2020 ~ '2010s',
ano >= 2020 ~ '2020s'
)) |>
group_by(decada, abbrev_state, name_region) |>
summarise(casos = sum(casos, na.rm = TRUE))
aids.dec
# A tibble: 135 × 4
# Groups: decada, abbrev_state [135]
decada abbrev_state name_region casos
<chr> <chr> <chr> <dbl>
1 1980s AC Norte 9
2 1980s AL Nordeste 0
3 1980s AM Norte 32
4 1980s AP Norte 7
5 1980s BA Nordeste 275
6 1980s CE Nordeste 117
7 1980s DF Centro Oeste 153
8 1980s ES Sudeste 117
9 1980s GO Centro Oeste 187
10 1980s MA Nordeste 55
# ℹ 125 more rows
# ℹ Use `print(n = ...)` to see more rowsAgora veja como fazer o boxplot:
ggplot(aids.dec, aes(decada, casos, fill = name_region)) +
geom_boxplot() +
theme_light()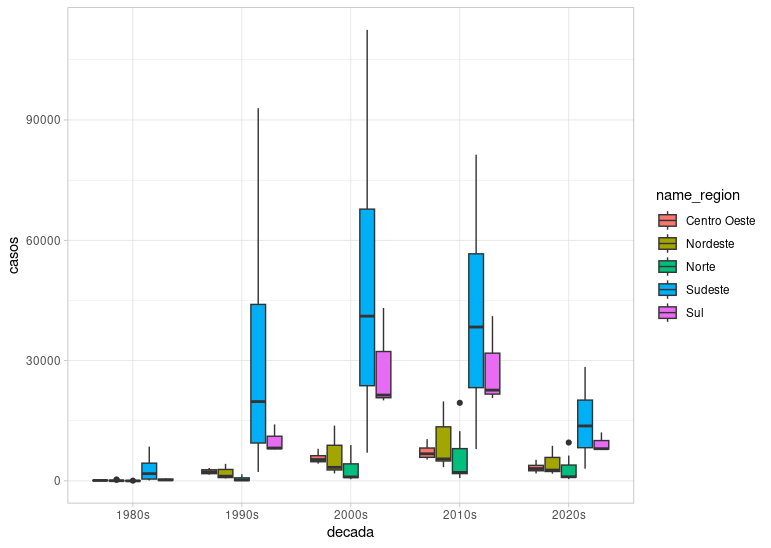
Poderíamos separar facetas por região:
ggplot(aids.dec, aes(decada, casos, fill = name_region)) +
geom_boxplot() +
facet_wrap(~name_region) +
theme_light() +
theme(legend.position = "none")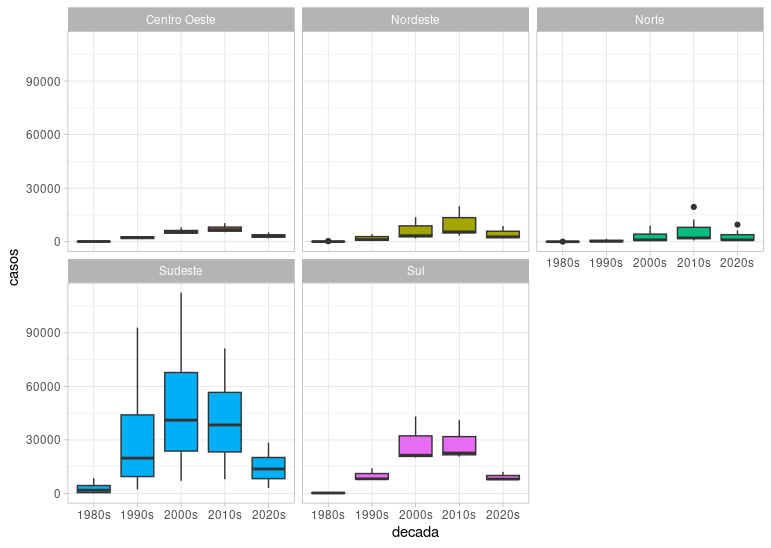
Que alterações você faria neste gráfico?
5.8 Gráficos de linha
O gráfico de linha é um dos tipos mais comuns de gráfico. Na epidemiologia o utilizamos muito para mostrar séries temporais de casos, incidência, etc. No ggplot, o gráfico de linha pode ser feito acrescentado a camada geom_line().
Podemos fazer uma série temporal de casos de AIDS no Brasil de 1980 a 2024.
url <- "https://raw.githubusercontent.com/laispfreitas/curso_CD1/refs/heads/main/aids_br.csv"
aids.br <- read_csv(url)
aids.br
# A tibble: 44 × 2
ano casos
<dbl> <dbl>
1 1980 1
2 1982 17
3 1983 42
4 1984 136
5 1985 535
6 1986 1121
7 1987 2708
8 1988 4353
9 1989 6019
10 1990 8660
# ℹ 34 more rows
# ℹ Use `print(n = ...)` to see more rowsVamos ao gráfico:
ggplot(aids.br, aes(x = ano, y = casos, color = casos)) +
geom_line(linewidth = 1.5) +
scale_color_continuous(type = "viridis") +
labs(title = "Total de casos de AIDS por ano, Brasil, 1980 a 2024*",
caption = "2024: dados preliminares*",
y = "Número de casos",
x = "Ano",
color = "Casos") +
ggthemes::theme_clean() 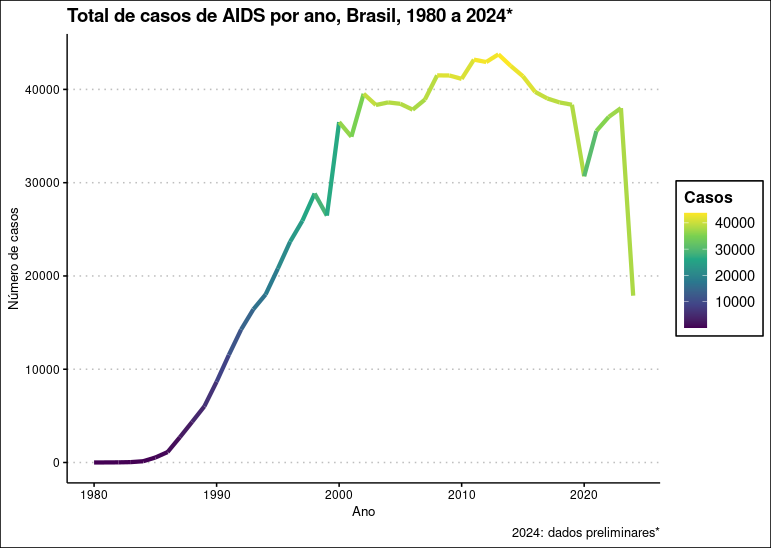
Perceba que associamos uma paleta à variável especificada em color = com scale_color_continuous()!
Podemos combinar geom_line() com geom_point():
ggplot(aids.br, aes(x = ano, y = casos, color = casos)) +
geom_line(linewidth = 1.5) +
geom_point(size = 3) +
scale_color_continuous(type = "viridis") +
labs(title = "Total de casos de AIDS por ano, Brasil, 1980 a 2024*",
caption = "2024: dados preliminares*",
y = "Número de casos",
x = "Ano",
color = "Casos") +
ggthemes::theme_clean() 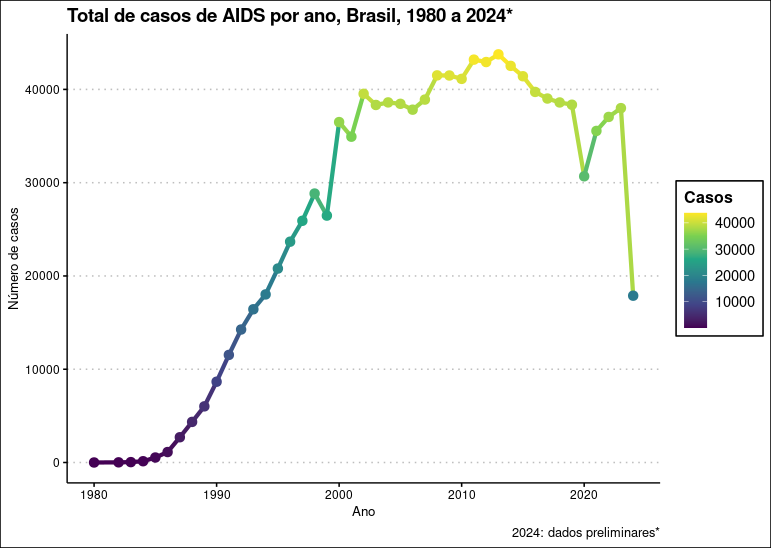
ATENÇÃO!!!
O geom_line() vai sempre criar um pedaço de linha entre as observações. Ou seja, se existirem dados faltantes e isso não for corrigido no dado, o gráfico ficará errado!!!
Vamos fazer o gráfico sem os ponto dos anos 2020 a 2023:
ggplot(aids.br |> filter(ano < 2020 | ano > 2023), aes(x = ano, y = casos, color = casos)) +
geom_line(linewidth = 1.5) +
geom_point(size = 3) +
scale_color_continuous(type = "viridis") +
ggthemes::theme_clean() 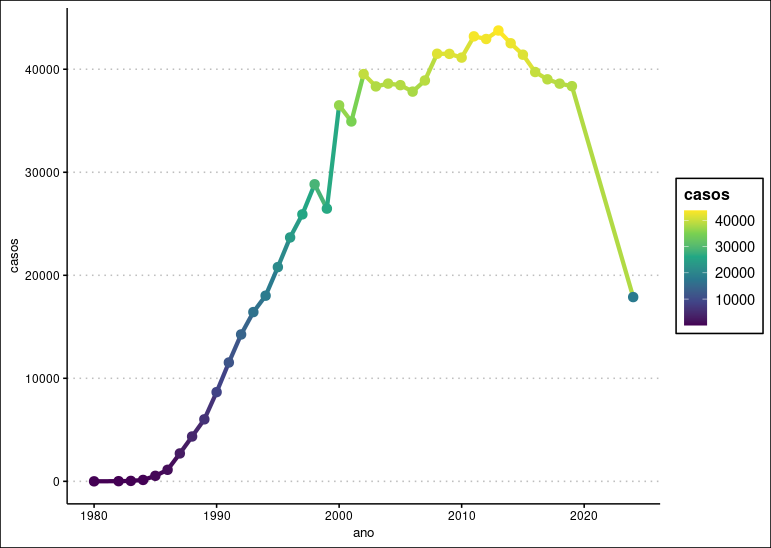
Repare no que aconteceu!
5.9 Mapas
É possível fazer mapas com o ggplot carregando também o pacote sf e acrescentando a camada geom_sf().
Vamos fazer um exemplo com os dados de um estudo sobre arboviroses e microcefalia em Pernambuco de 2013 a 2017 (Freitas L et al., 2023).
url <- "https://raw.githubusercontent.com/laispfreitas/PE_satscan/refs/heads/main/data_by_year.csv"
PE <- read_csv(url)
PE
# A tibble: 920 × 8
ID_MUNICIP year Population dengue zika chikungunya microcephaly live_births
<dbl> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl>
1 260005 2013 96769. 14 0 0 0 1532
2 260005 2014 96831. 23 0 0 0 1526
3 260005 2015 96907. 54 0 0 5 1628
4 260005 2016 96880. 23 0 29 2 1487
5 260005 2017 96828. 0 0 10 0 1519
6 260010 2013 36182. 0 0 0 0 638
7 260010 2014 36266. 5 0 0 0 653
8 260010 2015 36355. 891 0 39 2 666
9 260010 2016 36431. 205 0 23 3 580
10 260010 2017 36495. 24 0 8 0 522
# ℹ 910 more rows
# ℹ Use `print(n = ...)` to see more rowsEste dado é espaço-temporal. Para começar, vamos calcular a taxa de microcefalia por nascidos vivos para todo o período de estudo.
PE_total <- PE |>
group_by(ID_MUNICIP) |>
summarise(across(dengue:live_births, \(x) sum(x, na.rm = TRUE)),
pop = mean(Population)) |>
mutate(tx_micro = (microcephaly/live_births)*10000)
PE_total
# A tibble: 184 × 8
ID_MUNICIP dengue zika chikungunya microcephaly live_births pop tx_micro
<dbl> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl>
1 260005 114 0 39 7 7692 96843. 9.10
2 260010 1125 0 70 5 3059 36345. 16.3
3 260020 109 0 0 2 1415 18966. 14.1
4 260030 316 1 6 4 1736 24062. 23.0
5 260040 60 0 4 0 2565 35908. 0
6 260050 996 0 200 5 3724 42542. 13.4
7 260060 25 0 0 0 909 14434. 0
8 260070 398 0 47 6 2701 37825. 22.2
9 260080 523 0 563 2 1275 22411. 15.7
10 260090 34 0 16 0 1632 22543. 0
# ℹ 174 more rows
# ℹ Use `print(n = ...)` to see more rowsVamos precisar de um objeto sf com a geometria dos municípios de Pernambuco. Podemos obter com o pacote geobr:
library(sf)
library(geobr)
PE.sf <- read_municipality(code_muni = 26)
PE.sf
Simple feature collection with 185 features and 4 fields
Geometry type: MULTIPOLYGON
Dimension: XY
Bounding box: xmin: -41.35834 ymin: -9.48269 xmax: -32.39111 ymax: -3.828784
Geodetic CRS: SIRGAS 2000
First 10 features:
code_muni name_muni code_state abbrev_state geom
1 2600054 Abreu E Lima 26 PE MULTIPOLYGON (((-34.89651 -...
2 2600104 Afogados Da Ingazeira 26 PE MULTIPOLYGON (((-37.6847 -7...
3 2600203 Afrânio 26 PE MULTIPOLYGON (((-40.90285 -...
4 2600302 Agrestina 26 PE MULTIPOLYGON (((-35.96779 -...
5 2600401 Água Preta 26 PE MULTIPOLYGON (((-35.3699 -8...
6 2600500 Águas Belas 26 PE MULTIPOLYGON (((-36.98842 -...
7 2600609 Alagoinha 26 PE MULTIPOLYGON (((-36.82263 -...
8 2600708 Aliança 26 PE MULTIPOLYGON (((-35.16946 -...
9 2600807 Altinho 26 PE MULTIPOLYGON (((-36.02505 -...
10 2600906 Amaraji 26 PE MULTIPOLYGON (((-35.50934 -...Para juntar os dois objetos, vamos precisar criar o código do município de 6 dígitos em PE.sf.
Mas primeiro, vamos visualizar esse objeto:
ggplot() +
geom_sf(data = PE.sf) 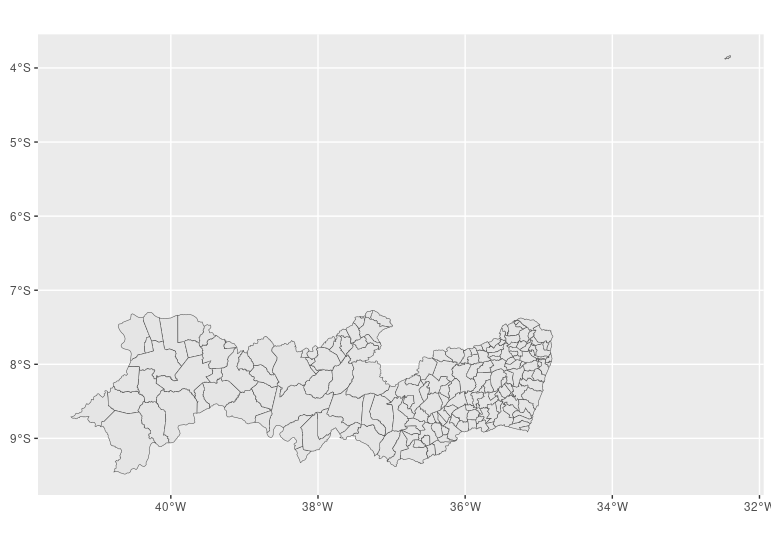
A ilha afastada é Fernando de Noronha, que não existe nos dados PE e que vai dificultar a visualização dos dados agora. Então, por ora, vamos excluí-la de PE.sf.
PE.sf <- PE.sf |>
mutate(ID_MUNICIP = str_sub(code_muni, 1, 6)) |>
filter(code_muni != 2605459)
PE_total <- PE_total |>
mutate(ID_MUNICIP = as.character(ID_MUNICIP))Agora vamos juntar os dois objetos. Neste caso, a ordem importa!
Veja o que acontece se fizermos:
PE_total |>
left_join(PE.sf, by = join_by(ID_MUNICIP)) |>
class()
[1] "tbl_df" "tbl" "data.frame"Agora veja o que acontece se fizermos:
PE.sf |>
left_join(PE_total, by = join_by(ID_MUNICIP)) |>
class()
[1] "sf" "data.frame"Portanto, como queremos fazer mapas, temos que fazer a ordem sf |> left_join(dataframe)!
PE.sf.casos <- PE.sf |>
left_join(PE_total, by = join_by(ID_MUNICIP))
PE.sf.casos
Simple feature collection with 184 features and 12 fields
Geometry type: MULTIPOLYGON
Dimension: XY
Bounding box: xmin: -41.35834 ymin: -9.48269 xmax: -34.80743 ymax: -7.272954
Geodetic CRS: SIRGAS 2000
First 10 features:
code_muni name_muni code_state abbrev_state ID_MUNICIP dengue zika chikungunya microcephaly live_births pop
1 2600054 Abreu E Lima 26 PE 260005 114 0 39 7 7692 96842.74
2 2600104 Afogados Da Ingazeira 26 PE 260010 1125 0 70 5 3059 36345.46
3 2600203 Afrânio 26 PE 260020 109 0 0 2 1415 18965.87
4 2600302 Agrestina 26 PE 260030 316 1 6 4 1736 24061.54
5 2600401 Água Preta 26 PE 260040 60 0 4 0 2565 35908.20
6 2600500 Águas Belas 26 PE 260050 996 0 200 5 3724 42542.15
7 2600609 Alagoinha 26 PE 260060 25 0 0 0 909 14433.64
8 2600708 Aliança 26 PE 260070 398 0 47 6 2701 37824.76
9 2600807 Altinho 26 PE 260080 523 0 563 2 1275 22411.03
10 2600906 Amaraji 26 PE 260090 34 0 16 0 1632 22543.40
tx_micro geom
1 9.100364 MULTIPOLYGON (((-34.89651 -...
2 16.345211 MULTIPOLYGON (((-37.6847 -7...
3 14.134276 MULTIPOLYGON (((-40.90285 -...
4 23.041475 MULTIPOLYGON (((-35.96779 -...
5 0.000000 MULTIPOLYGON (((-35.3699 -8...
6 13.426423 MULTIPOLYGON (((-36.98842 -...
7 0.000000 MULTIPOLYGON (((-36.82263 -...
8 22.213995 MULTIPOLYGON (((-35.16946 -...
9 15.686275 MULTIPOLYGON (((-36.02505 -...
10 0.000000 MULTIPOLYGON (((-35.50934 -...Agora vamos ao mapa!
ggplot() +
geom_sf(data = PE.sf.casos, aes(fill = tx_micro))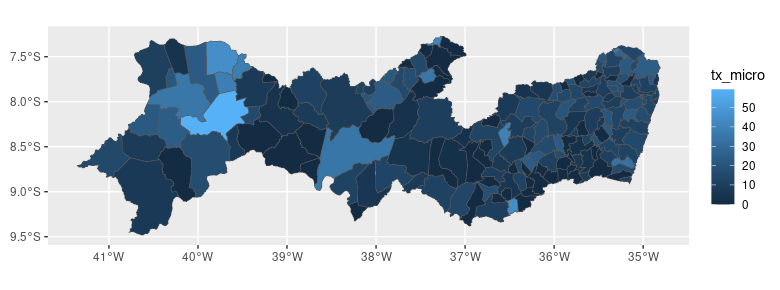
O ggplot2 automaticamente designou uma paleta de cores para o mapa. Ela é ideal?
Existem diversos pacotes de paletas para utilizar com o ggplot2. Dentre elas, descatam-se colorspace e RColorBrewer (saiba mais clicando aqui e aqui).
Vamos usar a colorspace. Ela possui funções “primas” das funções scale_* nativas do ggplot2:
library(colorspace)
g1 <- ggplot() +
geom_sf(data = PE.sf.casos, aes(fill = tx_micro)) +
scale_fill_continuous_sequential(palette = 'Heat',
name = "Microcefalia\npor 10mil") +
theme_void()
g1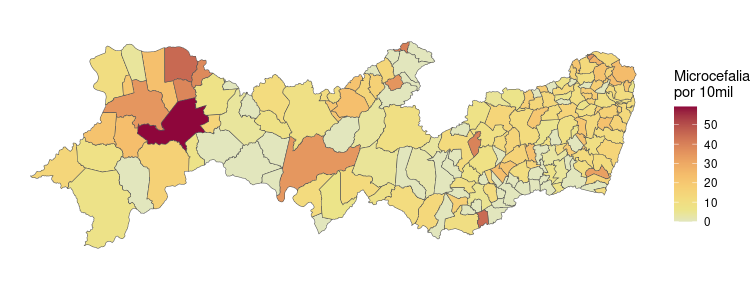
Agora, vamos fazer o mapa usando número de casos de microcefalia, e ver se a distribuição espacial muda. Vamos colocar os dois mapas na mesma figura usando a função ggarrange() do pacote ggpubr.
library(ggpubr)
g2 <- ggplot() +
geom_sf(data = PE.sf.casos, aes(fill = microcephaly)) +
scale_fill_continuous_sequential(palette = 'Heat',
name = "Microcefalia") +
theme_void()
ggarrange(g1, g2, nrow = 2, common.legend = FALSE)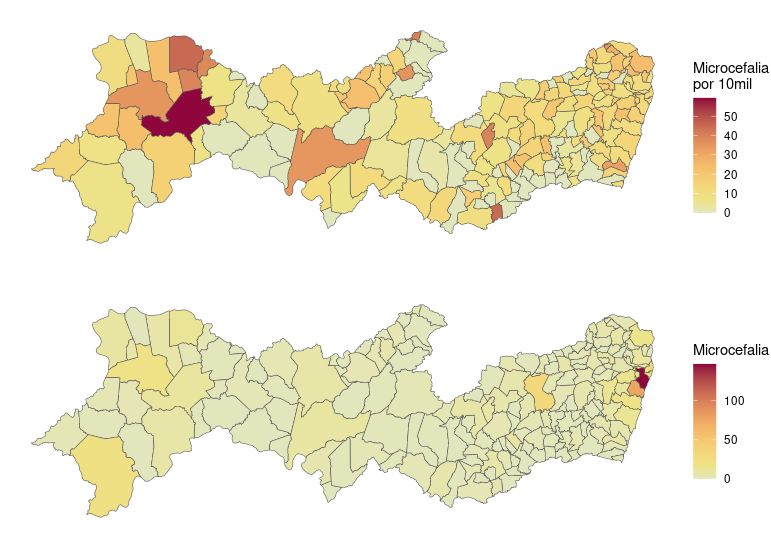
Qual mapa você escolheria? Por que?
Podemos usar mais de uma camada geom_sf() em um mapa. Vamos para isso fazer um mapa do número de casos de AIDS no Brasil por Unidade Federativa no ano de 2023. Vamos precisar dos polígonos dos estados e das regiões:
uf.sf <- read_state()
reg.sf <- read_region()
aids.sf <- uf.sf |>
left_join(aids.uf |> filter(ano == 2023),
by = join_by(code_state == cod_UF_res, abbrev_state, name_state, code_region, name_region))
aids.sf
Simple feature collection with 27 features and 7 fields
Geometry type: MULTIPOLYGON
Dimension: XY
Bounding box: xmin: -73.99045 ymin: -33.75208 xmax: -28.83594 ymax: 5.271841
Geodetic CRS: SIRGAS 2000
First 10 features:
code_state abbrev_state name_state code_region name_region ano casos geom
1 11 RO Rondônia 1 Norte 2023 403 MULTIPOLYGON (((-63.32721 -...
2 12 AC Acre 1 Norte 2023 144 MULTIPOLYGON (((-73.18253 -...
3 13 AM Amazonas 1 Norte 2023 1386 MULTIPOLYGON (((-67.32609 2...
4 14 RR Roraima 1 Norte 2023 271 MULTIPOLYGON (((-60.20051 5...
5 15 PA Pará 1 Norte 2023 2300 MULTIPOLYGON (((-54.95431 2...
6 16 AP Amapá 1 Norte 2023 211 MULTIPOLYGON (((-51.1797 4....
7 17 TO Tocantins 1 Norte 2023 276 MULTIPOLYGON (((-48.35878 -...
8 21 MA Maranhão 2 Nordeste 2023 1314 MULTIPOLYGON (((-45.84073 -...
9 22 PI Piauí 2 Nordeste 2023 422 MULTIPOLYGON (((-41.74605 -...
10 23 CE Ceará 2 Nordeste 2023 1525 MULTIPOLYGON (((-41.16703 -...Agora vamos pro mapa! Primeiro, vamos simplesmente fazer o mapa com a camada dos casos:
ggplot() +
geom_sf(data = aids.sf, aes(fill = casos)) +
scale_fill_continuous_sequential(palette = "Inferno", name = "Nº de casos") +
theme_void() 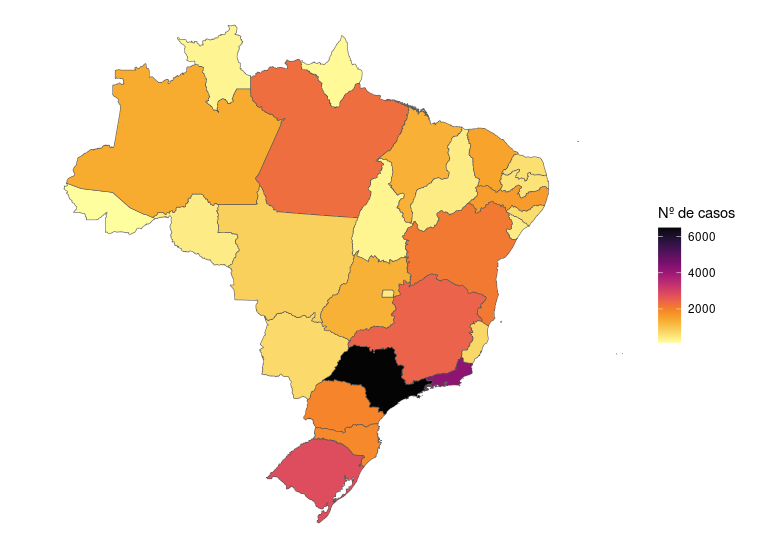
Agora vamos acrescentar a camada com a região:
ggplot() +
geom_sf(data = aids.sf, aes(fill = casos)) +
scale_fill_continuous_sequential(palette = "Inferno", name = "Nº de casos") +
geom_sf(data = reg.sf) +
theme_void() 
O que aconteceu?
As camadas aparecem na ordem que estão no código! Então a cor de preenchimento padrão de geom_sf(data = reg.sf) sobrepôs a escala de cores dos números de casos. Queremos somente as bordas das regiões, então precisamos especificar que não queremos preenchimento com fill = NA.
ggplot() +
geom_sf(data = aids.sf, aes(fill = casos)) +
scale_fill_continuous_sequential(palette = "Inferno", name = "Nº de casos") +
geom_sf(data = reg.sf, fill = NA) +
theme_void() 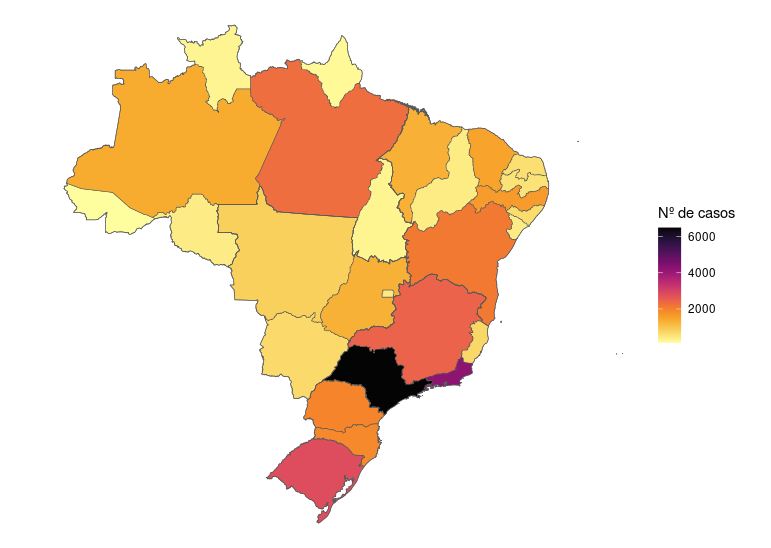
Agora vemos a escala de cores que está abaixo da camada de regiões, mas as bordas das regiões ainda não está muito distinta das bordas das UFs. Vamos mexer em alguns parâmetros pra melhorar a visualização deste mapa!
ggplot() +
geom_sf(data = aids.sf, aes(fill = casos), linewidth = .1, color = 'white') +
scale_fill_continuous_sequential(palette = "Inferno", name = "Nº de casos") +
geom_sf(data = reg.sf, fill = NA, color = 'grey20', linewidth = .5) +
labs(title = 'Número de casos de AIDS por Unidade Federativa, 2023') +
theme_void() +
theme(plot.title = element_text(hjust = .5, face = 'bold'))
5.10 Exercícios
- Importe os dados de temperatura por bairro e semana do Rio de Janeiro em 2016 (Freitas L et al., 2021):
- Verifique a distribuição da temperatura através de um histograma.
- Faça um boxplot da temperatura por semana.
- Obtenha o valor médio da temperatura por semana para o Rio de Janeiro e faça um gráfico de linha com a data no eixo x (dica: você vai precisar do pacote
aweek).
- Usando os dados de Pernambuco:
- Tente fazer mapas das incidências de dengue, Zika e chikungunya (um mapa para cada doença) e apresente-os em uma mesma figura.
5.11 Referências
Freitas LP, Lowe R, Koepp AE, Valongueiro SA, Dondero M, Marteleto LJ. “Identifying hidden Zika hotspots in Pernambuco, Brazil: A spatial analysis”. Transactions of the Royal Society of Tropical Medicine and Hygiene, 117(3):189–196 (2023). https://doi.org/10.1093/trstmh/trac099
Freitas LP, Schmidt AM, Cossich W, Cruz OG, Carvalho MS. “Spatio-temporal modelling of the first Chikungunya epidemic in an intra-urban setting: The role of socioeconomic status, environment and temperature”. PLOS Neglected Tropical Diseases, 15(6):e0009537 (2021). https://doi.org/10.1371/journal.pntd.0009537